+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10957 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of the Caulobacter crescentus flagellar motor and visualization of binding of a CheY-homolog | |||||||||
 マップデータ マップデータ | Flagellar motor of Caulobactor crescentus CB 15, in situ, cheYs deletion pdeA deletion pMT375 CleD-GFP, C100 symmetrised | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Caulobacter vibrioides CB15 (バクテリア) Caulobacter vibrioides CB15 (バクテリア) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 110.0 Å | |||||||||
 データ登録者 データ登録者 | Rossmann FM / Hug I / Sangermani M / Jenal U / Beeby M | |||||||||
| 資金援助 |  ドイツ, ドイツ,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Microbiol / 年: 2020 ジャーナル: Mol Microbiol / 年: 2020タイトル: In situ structure of the Caulobacter crescentus flagellar motor and visualization of binding of a CheY-homolog. 著者: Florian M Rossmann / Isabelle Hug / Matteo Sangermani / Urs Jenal / Morgan Beeby /   要旨: Bacterial flagellar motility is controlled by the binding of CheY proteins to the cytoplasmic switch complex of the flagellar motor, resulting in changes in swimming speed or direction. Despite its ...Bacterial flagellar motility is controlled by the binding of CheY proteins to the cytoplasmic switch complex of the flagellar motor, resulting in changes in swimming speed or direction. Despite its importance for motor function, structural information about the interaction between effector proteins and the motor are scarce. To address this gap in knowledge, we used electron cryotomography and subtomogram averaging to visualize such interactions inside Caulobacter crescentus cells. In C. crescentus, several CheY homologs regulate motor function for different aspects of the bacterial lifestyle. We used subtomogram averaging to image binding of the CheY family protein CleD to the cytoplasmic Cring switch complex, the control center of the flagellar motor. This unambiguously confirmed the orientation of the motor switch protein FliM and the binding of a member of the CheY protein family to the outside rim of the C ring. We also uncovered previously unknown structural elaborations of the alphaproteobacterial flagellar motor, including two novel periplasmic ring structures, and the stator ring harboring eleven stator units, adding to our growing catalog of bacterial flagellar diversity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10957.map.gz emd_10957.map.gz | 54.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10957-v30.xml emd-10957-v30.xml emd-10957.xml emd-10957.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10957.png emd_10957.png | 65.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10957 http://ftp.pdbj.org/pub/emdb/structures/EMD-10957 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10957 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10957 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10957_validation.pdf.gz emd_10957_validation.pdf.gz | 232.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10957_full_validation.pdf.gz emd_10957_full_validation.pdf.gz | 231.6 KB | 表示 | |
| XML形式データ |  emd_10957_validation.xml.gz emd_10957_validation.xml.gz | 6.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10957 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10957 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10957 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10957 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10957.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10957.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
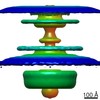
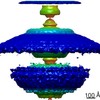
| 注釈 | Flagellar motor of Caulobactor crescentus CB 15, in situ, cheYs deletion pdeA deletion pMT375 CleD-GFP, C100 symmetrised | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.00206 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
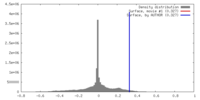
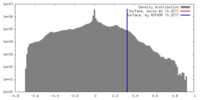
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Flagellar motor of Caulobactor crescentus CB 15, in situ, cheYs d...
| 全体 | 名称: Flagellar motor of Caulobactor crescentus CB 15, in situ, cheYs deletion pdeA deletion pMT375 CleD-GFP, C100 symmetrised |
|---|---|
| 要素 |
|
-超分子 #1: Flagellar motor of Caulobactor crescentus CB 15, in situ, cheYs d...
| 超分子 | 名称: Flagellar motor of Caulobactor crescentus CB 15, in situ, cheYs deletion pdeA deletion pMT375 CleD-GFP, C100 symmetrised タイプ: cell / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides CB15 (バクテリア) Caulobacter vibrioides CB15 (バクテリア) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 構成要素 - 名称: peptone yeast extract; PYE medium |
|---|---|
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.02 kPa |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: blot time 5 s, blot force 3, wait time 60s, drain time 0.5s,. |
| 詳細 | Flagellar motor of Caulobactor crescentus CB 15, in situ, cheYs deletion pdeA deletion pMT375 CleD-GFP, C100 symmetrised |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 7 / 平均露光時間: 1.5 sec. / 平均電子線量: 3.15 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 3.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 914 HIGH TILT LIQUID NITROGEN CRYO TRANSFER TOMOGRAPHY HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C100 (100回回転対称) / アルゴリズム: SIMULTANEOUS ITERATIVE (SIRT) / 解像度のタイプ: BY AUTHOR / 解像度: 110.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: PEET (ver. 1.10.1) / 使用したサブトモグラム数: 114 |
|---|---|
| 抽出 | トモグラム数: 131 / 使用した粒子像数: 114 / 参照モデル: sperical / 手法: manual / ソフトウェア - 名称: PEET (ver. 1.10.1) |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: PEET (ver. 1.10.1) |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)