+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10658 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the ring-shaped virulence factor EspB from Mycobacterium tuberculosis | |||||||||
 マップデータ マップデータ | Sharped map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | M. tuberculosis / ESX-1 / Type VII secretion system / EspB / Rv3881c / EsxA / CFP10 / ESAT-6 / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 | ESX-1 secretion-associated protein EspB, PE domain / ESX-1 secreted protein B PE domain / protein secretion by the type VII secretion system / PPE superfamily / biological process involved in interaction with host / extracellular region / identical protein binding / ESX-1 secretion-associated protein EspB 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Mycobacterium tuberculosis H37Rv (結核菌) / Mycobacterium tuberculosis H37Rv (結核菌) /  Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.37 Å | |||||||||
 データ登録者 データ登録者 | Piton J / Pojer F | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Struct Biol X / 年: 2020 ジャーナル: J Struct Biol X / 年: 2020タイトル: High resolution CryoEM structure of the ring-shaped virulence factor EspB from . 著者: Jérémie Piton / Florence Pojer / Soichi Wakatsuki / Cornelius Gati / Stewart T Cole /   要旨: The EspB protein of is a 60 kDa virulence factor, implicated in conjugation and exported by the ESX-1 system of which it may also be a component. Previous attempts to obtain high-resolution maps of ...The EspB protein of is a 60 kDa virulence factor, implicated in conjugation and exported by the ESX-1 system of which it may also be a component. Previous attempts to obtain high-resolution maps of EspB by cryo-electron microscopic examination of single particles have been thwarted by severe orientation bias of the particles. This was overcome by using detergent as a surfactant thereby allowing reconstruction of the EspB structure at 3.37 Å resolution. The final structure revealed the N-terminal domain of EspB to be organized as a cylindrical heptamer with dimensions of 90 Å x 90 Å and a central channel of 45 Å diameter whereas the C-terminal domain was unstructured. New atomic insight was obtained into the helical packing required for protomer interactions and the overall electrostatic potential. The external surface is electronegatively charged while the channel is lined with electropositive patches. EspB thus has many features of a pore-like transport protein that might allow the passage of an ESX-1 substrate such as the 35 Å diameter EsxA-EsxB heterodimer or B-form DNA consistent with its proposed role in DNA uptake. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10658.map.gz emd_10658.map.gz | 230.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10658-v30.xml emd-10658-v30.xml emd-10658.xml emd-10658.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10658.png emd_10658.png | 58 KB | ||
| マスクデータ |  emd_10658_msk_1.map emd_10658_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10658.cif.gz emd-10658.cif.gz | 6.3 KB | ||
| その他 |  emd_10658_half_map_1.map.gz emd_10658_half_map_1.map.gz emd_10658_half_map_2.map.gz emd_10658_half_map_2.map.gz | 226.5 MB 226.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10658 http://ftp.pdbj.org/pub/emdb/structures/EMD-10658 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10658 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10658 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10658_validation.pdf.gz emd_10658_validation.pdf.gz | 1017.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10658_full_validation.pdf.gz emd_10658_full_validation.pdf.gz | 1017 KB | 表示 | |
| XML形式データ |  emd_10658_validation.xml.gz emd_10658_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_10658_validation.cif.gz emd_10658_validation.cif.gz | 19.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10658 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10658 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10658 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10658 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10658.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10658.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharped map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.058 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10658_msk_1.map emd_10658_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
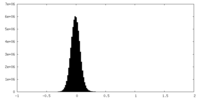
| 密度ヒストグラム |
-ハーフマップ: Map A
| ファイル | emd_10658_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
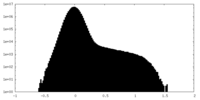
| 密度ヒストグラム |
-ハーフマップ: Map B
| ファイル | emd_10658_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EspB Heptameric complex
| 全体 | 名称: EspB Heptameric complex |
|---|---|
| 要素 |
|
-超分子 #1: EspB Heptameric complex
| 超分子 | 名称: EspB Heptameric complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) |
| 分子量 | 理論値: 329 KDa |
-分子 #1: ESX-1 secretion-associated protein EspB
| 分子 | 名称: ESX-1 secretion-associated protein EspB / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌)株: ATCC 25618 / H37Rv |
| 分子量 | 理論値: 47.637527 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTQSQTVTVD QQEILNRANE VEAPMADPPT DVPITPCELT AAKNAAQQLV LSADNMREYL AAGAKERQRL ATSLRNAAKA YGEVDEEAA TALDNDGEGT VQAESAGAVG GDSSAELTDT PRVATAGEPN FMDLKEAARK LETGDQGASL AHFADGWNTF N LTLQGDVK ...文字列: MTQSQTVTVD QQEILNRANE VEAPMADPPT DVPITPCELT AAKNAAQQLV LSADNMREYL AAGAKERQRL ATSLRNAAKA YGEVDEEAA TALDNDGEGT VQAESAGAVG GDSSAELTDT PRVATAGEPN FMDLKEAARK LETGDQGASL AHFADGWNTF N LTLQGDVK RFRGFDNWEG DAATACEASL DQQRQWILHM AKLSAAMAKQ AQYVAQLHVW ARREHPTYED IVGLERLYAE NP SARDQIL PVYAEYQQRS EKVLTEYNNK AALEPVNPPK PPPAIKIDPP PPPQEQGLIP GFLMPPSDGS GVTPGTGMPA APM VPPTGS PGGGLPADTA AQLTSAGREA AALSGDVAVK AASLGGGGGG GVPSAPLGSA IGGAESVRPA GAGDIAGLGQ GRAG GGAAL GGGGMGMPMG AAHQGQGGAK SKGSQQEDEA LYTEDRAWTE AVIGNRRRQD SKESK UniProtKB: ESX-1 secretion-associated protein EspB |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. | ||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV 詳細: 0.05% fluorinated octylmaltoside was add into protein solution and immediately applied to a freshly glow-discharged holey carbon grid. ). Excess liquid was blotted for 2.3 s using an FEI ...詳細: 0.05% fluorinated octylmaltoside was add into protein solution and immediately applied to a freshly glow-discharged holey carbon grid. ). Excess liquid was blotted for 2.3 s using an FEI Vitrobot Mark IV and the sample was plunge frozen in liquid ethane.. | ||||||
| 詳細 | This sample was monodiperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 2600 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X