+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10087 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Acquired functional capsid structures in metazoan totivirus-like dsRNA virus. | ||||||||||||
 マップデータ マップデータ | Omono River virus capsid | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Mosquito / Totivirus / Totiviridae / Totivirus-like virus / dsRNA / capsid / VIRUS | ||||||||||||
| 機能・相同性 | Double-stranded RNA binding motif / Double-stranded RNA binding motif / Double stranded RNA-binding domain (dsRBD) profile. / Double-stranded RNA-binding domain / RNA binding / Capsid protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Omono River virus (ウイルス) Omono River virus (ウイルス) | ||||||||||||
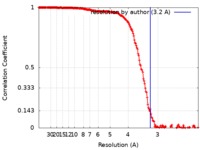
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||
 データ登録者 データ登録者 | Okamoto K / Larsson SDD | ||||||||||||
| 資金援助 |  スウェーデン, 3件 スウェーデン, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2020 ジャーナル: Structure / 年: 2020タイトル: Acquired Functional Capsid Structures in Metazoan Totivirus-like dsRNA Virus. 著者: Kenta Okamoto / Ricardo J Ferreira / Daniel S D Larsson / Filipe R N C Maia / Haruhiko Isawa / Kyoko Sawabe / Kazuyoshi Murata / Janos Hajdu / Kenji Iwasaki / Peter M Kasson / Naoyuki Miyazaki /    要旨: Non-enveloped icosahedral double-stranded RNA (dsRNA) viruses possess multifunctional capsids required for their proliferation. Whereas protozoan/fungal dsRNA viruses have a relatively simple capsid ...Non-enveloped icosahedral double-stranded RNA (dsRNA) viruses possess multifunctional capsids required for their proliferation. Whereas protozoan/fungal dsRNA viruses have a relatively simple capsid structure, which suffices for the intracellular phase in their life cycle, metazoan dsRNA viruses have acquired additional structural features as an adaptation for extracellular cell-to-cell transmission in multicellular hosts. Here, we present the first atomic model of a metazoan dsRNA totivirus-like virus and the structure reveals three unique structural traits: a C-terminal interlocking arm, surface projecting loops, and an obstruction at the pore on the 5-fold symmetry axis. These traits are keys to understanding the capsid functions of metazoan dsRNA viruses, such as particle stability and formation, cell entry, and endogenous intraparticle transcription of mRNA. On the basis of molecular dynamics simulations of the obstructed pore, we propose a possible mechanism of intraparticle transcription in totivirus-like viruses, which dynamically switches between open and closed states of the pore(s). | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10087.map.gz emd_10087.map.gz | 211 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10087-v30.xml emd-10087-v30.xml emd-10087.xml emd-10087.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10087_fsc.xml emd_10087_fsc.xml | 20.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10087.png emd_10087.png | 293.5 KB | ||
| Filedesc metadata |  emd-10087.cif.gz emd-10087.cif.gz | 5.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10087 http://ftp.pdbj.org/pub/emdb/structures/EMD-10087 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10087 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10087 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10087_validation.pdf.gz emd_10087_validation.pdf.gz | 675.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10087_full_validation.pdf.gz emd_10087_full_validation.pdf.gz | 674.9 KB | 表示 | |
| XML形式データ |  emd_10087_validation.xml.gz emd_10087_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_10087_validation.cif.gz emd_10087_validation.cif.gz | 22.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10087 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10087 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10087 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10087 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10087.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10087.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Omono River virus capsid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Omono River virus
| 全体 | 名称:  Omono River virus (ウイルス) Omono River virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Omono River virus
| 超分子 | 名称: Omono River virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Omono River virus particles from C6/36 mosquito cells NCBI-ID: 753758 / 生物種: Omono River virus / Sci species strain: AK4 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 420.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Omono River virus (ウイルス) Omono River virus (ウイルス) |
| 分子量 | 理論値: 91.202719 KDa |
| 配列 | 文字列: PISADFSEVE NAPSFLSLAE NTDEVLKPYT GLEIQTIITN IVGDANPNQS RIFDQDRLRG NQYSAGGLVT QNAVSAIPFT NLIPRTIRV GNILVNSANR LQITETNVSE YYSNPIIATK LSEMISDQVK NNQFSTWRRD NTSLQGFNAF DIATINTAIL P NGLSLESM ...文字列: PISADFSEVE NAPSFLSLAE NTDEVLKPYT GLEIQTIITN IVGDANPNQS RIFDQDRLRG NQYSAGGLVT QNAVSAIPFT NLIPRTIRV GNILVNSANR LQITETNVSE YYSNPIIATK LSEMISDQVK NNQFSTWRRD NTSLQGFNAF DIATINTAIL P NGLSLESM LLKLSLLHSI KAMNVDAASI NRSQYQVIDH NTVPTIGAPA VVGVNNSPVF GEDCGGNNPV YPFGGGTGAI AF HVTLQTV PDERKSYAIF VPPAILQATS DANEALALFA LSMSEWPHAL YTVTKQTTDL AGANAGQQVF IPTQSTIHIG GRR VLDLII PRREIAPNPT TLVAANAMCM VRPQAGPDAT AGAIPLAAGQ LFNMNFIGAP AFEEWPLTSY LYSWAGRFDI TTIR QYMGR LATMVGVKDA YWAAHELNVA LSQVAPKMTT AAGGWAAQAA NSAQQSDVCY SSLLTVTRSA ANFPLANQPA ADMRV YDTD PATWNKVALG LATAANLVPE QSMDVPFVVG DARTSFWERL QAIPMCIAWT MYYHSRGITT LAWDNAYTDN TNKWLQ KMV RNTFSTTQSV GTIIPARYGK IVCNLYKNMF HRAPAYVATS VGGKELHITH FERWLPGGTY ANVYSGAGAV VNCFSPV LI PDIWCQYFTA KLPLFAGAFP PAQGQNSTKG FNSKQGLMIH RNQNNNLVAP YLEKFADNSS YFPVGQGPEI NDMATWNG R LWMTTGNVQY LDYSGAAIVE AVPPAGELPV GKQIPLLAGE NAPIELTNAA TTCVPRYSND GRRIFTYLTT AQSVIPVQA CNRAANLARS CWLLSNVYAE PALQALGDEV EDAFDTLTNS UniProtKB: Capsid protein |
-分子 #2: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Omono River virus (ウイルス) Omono River virus (ウイルス) |
| 分子量 | 理論値: 91.436969 KDa |
| 配列 | 文字列: PISADFSEVE NAPSFLSLAE NTDEVLKPYT GLEIQTIITN IVGDANPNQS RIFDQDRLRG NQYSAGGLVT QNAVSAIPFT NLIPRTIRV GNILVNSANR LQITETNVSE YYSNPIIATK LSEMISDQVK NNQFSTWRRD NTSLQGFNAF DIATINTAIL P NGLSLESM ...文字列: PISADFSEVE NAPSFLSLAE NTDEVLKPYT GLEIQTIITN IVGDANPNQS RIFDQDRLRG NQYSAGGLVT QNAVSAIPFT NLIPRTIRV GNILVNSANR LQITETNVSE YYSNPIIATK LSEMISDQVK NNQFSTWRRD NTSLQGFNAF DIATINTAIL P NGLSLESM LLKLSLLHSI KAMNVDAASI NRSQYQVIDH NTVPTIGAPA VVGVNNSPVF GEDCGGNNPV YPFGGGTGAI AF HVTLQTV PDERKSYAIF VPPAILQATS DANEALALFA LSMSEWPHAL YTVTKQTTDL AGANAGQQVF IPTQSTIHIG GRR VLDLII PRREIAPNPT TLVAANAMCM VRPQAGPDAT AGAIPLAAGQ LFNMNFIGAP AFEEWPLTSY LYSWAGRFDI TTIR QYMGR LATMVGVKDA YWAAHELNVA LSQVAPKMTT AAGGWAAQAA NSAQQSDVCY SSLLTVTRSA ANFPLANQPA ADMRV YDTD PATWNKVALG LATAANLVPE QSMDVPFVVG DARTSFWERL QAIPMCIAWT MYYHSRGITT LAWDNAYTDN TNKWLQ KMV RNTFSTTQSV GTIIPARYGK IVCNLYKNMF HRAPAYVATS VGGKELHITH FERWLPGGTY ANVYSGAGAV VNCFSPV LI PDIWCQYFTA KLPLFAGAFP PAQGQNSTKG FNSKQGLMIH RNQNNNLVAP YLEKFADNSS YFPVGQGPEI NDMATWNG R LWMTTGNVQY LDYSGAAIVE AVPPAGELPV GKQIPLLAGE NAPIELTNAA TTCVPRYSND GRRIFTYLTT AQSVIPVQA CNRAANLARS CWLLSNVYAE PALQALGDEV EDAFDTLTNS SF UniProtKB: Capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 100.0 mM / 構成要素 - 式: C2H7NO2 / 構成要素 - 名称: ammonium acetate |
| グリッド | 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: COUNTING / 実像数: 4521 / 平均露光時間: 1.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.225 µm / 最小 デフォーカス(公称値): 0.1 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6s2c: |
 ムービー
ムービー コントローラー
コントローラー