万見
万見+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1001 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Real space refinement of acto-myosin structures from sectioned muscle. | |||||||||
 マップデータ マップデータ | Acto-myosin structures from sectioned muscle | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of myosin II filament assembly / contractile muscle fiber / Striated Muscle Contraction / myosin filament / myosin II complex / myosin complex / cytoskeletal motor activator activity / microfilament motor activity / myofibril / tropomyosin binding ...regulation of myosin II filament assembly / contractile muscle fiber / Striated Muscle Contraction / myosin filament / myosin II complex / myosin complex / cytoskeletal motor activator activity / microfilament motor activity / myofibril / tropomyosin binding / myosin heavy chain binding / mesenchyme migration / troponin I binding / filamentous actin / actin filament bundle / skeletal muscle thin filament assembly / actin filament bundle assembly / striated muscle thin filament / skeletal muscle myofibril / actin monomer binding / skeletal muscle fiber development / stress fiber / titin binding / skeletal muscle tissue development / muscle contraction / actin filament polymerization / filopodium / actin filament / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / calcium-dependent protein binding / actin filament binding / lamellipodium / cell body / calmodulin binding / hydrolase activity / protein domain specific binding / calcium ion binding / positive regulation of gene expression / magnesium ion binding / ATP binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lethocerus (昆虫) Lethocerus (昆虫) | |||||||||
| 手法 | 電子線トモグラフィー法 / ネガティブ染色法 / 解像度: 40.0 Å | |||||||||
 データ登録者 データ登録者 | Chen LF / Blanc E / Chapman MS / Taylor KA | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2001 ジャーナル: J Struct Biol / 年: 2001タイトル: Real space refinement of acto-myosin structures from sectioned muscle. 著者: L F Chen / E Blanc / M S Chapman / K A Taylor /  要旨: We have adapted a real space refinement protocol originally developed for high-resolution crystallographic analysis for use in fitting atomic models of actin filaments and myosin subfragment 1 (S1) ...We have adapted a real space refinement protocol originally developed for high-resolution crystallographic analysis for use in fitting atomic models of actin filaments and myosin subfragment 1 (S1) to 3-D images of thin-sectioned, plastic-embedded whole muscle. The rationale for this effort is to obtain a refinement protocol that will optimize the fit of the model to the density obtained by electron microscopy and correct for poor geometry introduced during the manual fitting of a high-resolution atomic model into a lower resolution 3-D image. The starting atomic model consisted of a rigor acto-S1 model obtained by X-ray crystallography and helical reconstruction of electron micrographs. This model was rebuilt to fit 3-D images of rigor insect flight muscle at a resolution of 7 nm obtained by electron tomography and image averaging. Our highly constrained real space refinement resulted in modest improvements in the agreement of model and reconstruction but reduced the number of conflicting atomic contacts by 70% without loss of fit to the 3-D density. The methodology seems to be well suited to the derivation of stereochemically reasonable atomic models that are consistent with experimentally determined 3-D reconstructions computed from electron micrographs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1001.map.gz emd_1001.map.gz | 38 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1001-v30.xml emd-1001-v30.xml emd-1001.xml emd-1001.xml | 70.2 KB 70.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1001.gif 1001.gif | 60.3 KB | ||
| マスクデータ |  emd_1001_msk_1.map emd_1001_msk_1.map emd_1001_msk_10.map emd_1001_msk_10.map emd_1001_msk_11.map emd_1001_msk_11.map emd_1001_msk_12.map emd_1001_msk_12.map emd_1001_msk_13.map emd_1001_msk_13.map emd_1001_msk_14.map emd_1001_msk_14.map emd_1001_msk_15.map emd_1001_msk_15.map emd_1001_msk_16.map emd_1001_msk_16.map emd_1001_msk_17.map emd_1001_msk_17.map emd_1001_msk_18.map emd_1001_msk_18.map emd_1001_msk_19.map emd_1001_msk_19.map emd_1001_msk_2.map emd_1001_msk_2.map emd_1001_msk_20.map emd_1001_msk_20.map emd_1001_msk_21.map emd_1001_msk_21.map emd_1001_msk_22.map emd_1001_msk_22.map emd_1001_msk_23.map emd_1001_msk_23.map emd_1001_msk_24.map emd_1001_msk_24.map emd_1001_msk_25.map emd_1001_msk_25.map emd_1001_msk_26.map emd_1001_msk_26.map emd_1001_msk_3.map emd_1001_msk_3.map emd_1001_msk_4.map emd_1001_msk_4.map emd_1001_msk_5.map emd_1001_msk_5.map emd_1001_msk_6.map emd_1001_msk_6.map emd_1001_msk_7.map emd_1001_msk_7.map emd_1001_msk_8.map emd_1001_msk_8.map emd_1001_msk_9.map emd_1001_msk_9.map | 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 34.8 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB 136 KB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1001 http://ftp.pdbj.org/pub/emdb/structures/EMD-1001 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1001 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1001 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1001_validation.pdf.gz emd_1001_validation.pdf.gz | 274.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1001_full_validation.pdf.gz emd_1001_full_validation.pdf.gz | 273.7 KB | 表示 | |
| XML形式データ |  emd_1001_validation.xml.gz emd_1001_validation.xml.gz | 4.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1001 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1001 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1001 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1001 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1m8qMK  1mvwMK  1o18MK  1o19MK  1o1aMK  1o1bMK  1o1cMK  1o1dMK  1o1eMK  1o1fMK  1o1gMK M: このマップから作成された原子モデル K: マスクデータへのあてはめ*YM |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1001.map.gz / 形式: CCP4 / 大きさ: 40.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1001.map.gz / 形式: CCP4 / 大きさ: 40.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Acto-myosin structures from sectioned muscle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 15.4667 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
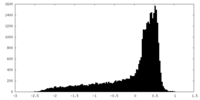
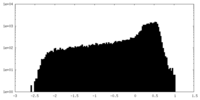
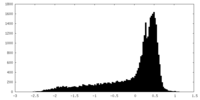
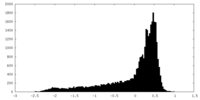
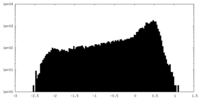
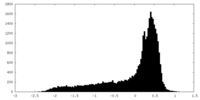
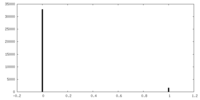
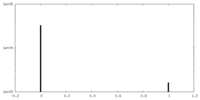
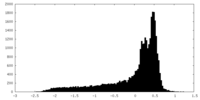
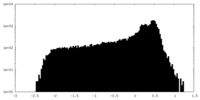
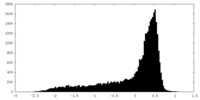
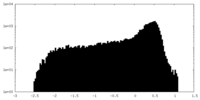
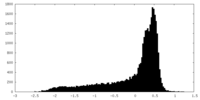
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
+セグメンテーションマップ: #2
+セグメンテーションマップ: #11
+セグメンテーションマップ: #12
+セグメンテーションマップ: #13
+セグメンテーションマップ: #14
+セグメンテーションマップ: #15
+セグメンテーションマップ: #16
+セグメンテーションマップ: #17
+セグメンテーションマップ: #18
+セグメンテーションマップ: #19
+セグメンテーションマップ: #20
+セグメンテーションマップ: #3
+セグメンテーションマップ: #21
+セグメンテーションマップ: #22
+セグメンテーションマップ: #23
+セグメンテーションマップ: #24
+セグメンテーションマップ: #25
+セグメンテーションマップ: #26
+セグメンテーションマップ: #1
+セグメンテーションマップ: #4
+セグメンテーションマップ: #5
+セグメンテーションマップ: #6
+セグメンテーションマップ: #7
+セグメンテーションマップ: #8
+セグメンテーションマップ: #9
+セグメンテーションマップ: #10
- 試料の構成要素
試料の構成要素
-全体 : Rigor insect flight muscle from Lethocerus maximus
| 全体 | 名称: Rigor insect flight muscle from Lethocerus maximus |
|---|---|
| 要素 |
|
-超分子 #1000: Rigor insect flight muscle from Lethocerus maximus
| 超分子 | 名称: Rigor insect flight muscle from Lethocerus maximus / タイプ: sample / ID: 1000 詳細: The sample is a single filament layer cut from myofibrils by ultramicrotomy. The section thickness is ~25 nm. Specimen has been chemically fixed, dehydrated, embedded in Araldite and ...詳細: The sample is a single filament layer cut from myofibrils by ultramicrotomy. The section thickness is ~25 nm. Specimen has been chemically fixed, dehydrated, embedded in Araldite and sectioned with a diamond knife. 集合状態: Thick myosin containing filament and thin actin-containing filaments. Number unique components: 2 |
|---|
-超分子 #1: thick, myosin-containing filament
| 超分子 | 名称: thick, myosin-containing filament / タイプ: organelle_or_cellular_component / ID: 1 / Name.synonym: myosin / 詳細: Filaments contain more than just myosin / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  Lethocerus (昆虫) / 別称: water bug, Lethocerus maximus / 組織: Indirect flight muscles / 細胞: muscle fibers / Organelle: myofibrils / 細胞中の位置: myofibrils Lethocerus (昆虫) / 別称: water bug, Lethocerus maximus / 組織: Indirect flight muscles / 細胞: muscle fibers / Organelle: myofibrils / 細胞中の位置: myofibrils |
-超分子 #2: thin, actin-containing filament
| 超分子 | 名称: thin, actin-containing filament / タイプ: organelle_or_cellular_component / ID: 2 / Name.synonym: actin / 詳細: filaments contain more than just actin 集合状態: polar, 2-stranded helical filament, helical symmetry is 28/13 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  Lethocerus (昆虫) / 別称: water bug, Lethocerus maximus / 組織: Indirect flight muscles / 細胞: Muscle fibers / Organelle: myofibrils / 細胞中の位置: myofibrils Lethocerus (昆虫) / 別称: water bug, Lethocerus maximus / 組織: Indirect flight muscles / 細胞: Muscle fibers / Organelle: myofibrils / 細胞中の位置: myofibrils |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
- 試料調製
試料調製
| 凍結 | 凍結剤: NONE |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS EM400 |
|---|---|
| 温度 | 最低: 25 K / 最高: 25 K / 平均: 25 K |
| 撮影 | デジタル化 - スキャナー: PERKIN ELMER / デジタル化 - サンプリング間隔: 25 µm / 実像数: 24 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 17000 |
| 試料ステージ | 試料ホルダー: Philips rotation-tilt holder / 試料ホルダーモデル: PHILIPS ROTATION HOLDER / Tilt series - Axis1 - Min angle: 4.3 ° / Tilt series - Axis1 - Max angle: 59.4 ° / Tilt series - Axis1 - Angle increment: 10 ° |
- 画像解析
画像解析
| 詳細 | Double axis tilt series collected on film. Digitized on PDS 1010M densitometer. Other relevent references K. A. Taylor, M. C. Reedy, L. Cordova and M. K. Reedy. 3-D structure of insect flight muscle in rigor from tilted thin sections. Nature 310, 28 5-291 (1984). Hanspeter Winkler and Kenneth A. Taylor. Multivariate statistical analysis of three-dimensional cross-bridge motifs in insect flight muscle. Ultramicroscopy 77, 141-152 (1999). Chen, Li Fan, Winkler, Hanspeter, Reedy, Michael K., Ree dy, Mary C. and Taylor, Kenneth A.. Molecular Modeling of Averaged Rigor Crossbridges from Tomograms of Insect Flight Muscle. J. Struct. Biol. 138(2), 92-104 (2002) |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 40.0 Å / 解像度の算出法: OTHER 詳細: The reconstruction was done by aligning the members of the tilt series using crosscorrelation functions, such as phase only and the VanHeel Mutual Correlation Function. The sampling in each ...詳細: The reconstruction was done by aligning the members of the tilt series using crosscorrelation functions, such as phase only and the VanHeel Mutual Correlation Function. The sampling in each micrographof a tilted specimen was match by interpolation to th e lower angle members of the tilt series. This matching of areas and sampling was done by a 4 parameter grid search. Once aligned and scaled, the data were combined in Fourier space using a Whittaker-Shannon interpolation scheme. The map was calculated using a reverse Fourier transformation. The software used for the reconstruction was in-house and adapted from in some cases from MRC software. 使用した粒子像数: 30 |
| CTF補正 | 詳細: none |
-原子モデル構築 1
| ソフトウェア | 名称: RSref |
|---|---|
| 詳細 | Protocol: real space rigid body. Domains were fit manually into the density using O. The entire light chain domain consisting of heavy chain residues from G710 to K843 and the two light chains were repositioned using G710 of the myosin heavy chain using O.Real space refinement done using 7 rigid bodies and 4 hinge points. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: poor atom-atom contacts and cross correlation coefficient |
| 得られたモデル |  PDB-1m8q:  PDB-1mvw:  PDB-1o18:  PDB-1o19:  PDB-1o1a:  PDB-1o1b:  PDB-1o1c:  PDB-1o1d:  PDB-1o1e:  PDB-1o1f:  PDB-1o1g: |
 ムービー
ムービー コントローラー
コントローラー


 UCSF Chimera
UCSF Chimera








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)