[日本語] English
 万見
万見- ChemComp-0GE: N-{[5-(dimethylamino)naphthalen-1-yl]sulfonyl}-L-alpha-glutamyl-N... -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 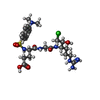 データベース: PDB化学物質要素 / ID: 0GE データベース: PDB化学物質要素 / ID: 0GE |
|---|---|
| 名称 | 名称: 別称: 1,5-DANSYL-GLU-GLY-ARG-CHLOROMETHYL KETONE, bound form |
-BIRD information
| タイプ | ペプチド様 / 阻害剤 |
|---|---|
 ダウンロード ダウンロード |  Molecular definition / Molecular definition /  Chemical definition / Chemical definition /  Family definition Family definition |
| 別称 |
|
| Annotation |
|
| External info |
|
| Family | EGR-chloromethylketone inhibitor dansyl-Glu-Gly-Arg chloromethyl ketone / GLU-GLY-ARG-CHLOROMETHYL KETONE / dansylglutamyl-glycyl-arginine chloromethyl ketone |
-Chemical information
| 組成 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| その他 | タイプ: peptide-like / PDB分類: HETAIN / 3文字コード: 0GE / 理論座標の詳細: Corina / モデル座標のPDB-ID: 1CVW / モデル座標の詳細: not provided / 子要素: ANS, GLU, GLY, AR7, 0QE | ||||||||||||
| 履歴 |
| ||||||||||||
 外部リンク 外部リンク |  UniChem / UniChem /  ChemSpider / ChemSpider /  PubChem / PubChem /  Wikipedia search / Wikipedia search /  Google search Google search |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
-詳細
-SMILES
| ACDLabs 12.01 | | CACTVS 3.370 | OpenEye OEToolkits 1.7.2 | |
|---|
-SMILES CANONICAL
| CACTVS 3.370 | | OpenEye OEToolkits 1.7.2 | [ | |
|---|
-InChI
| InChI 1.03 |
|---|
-InChIKey
| InChI 1.03 |
|---|
-SYSTEMATIC NAME
| ACDLabs 12.01 | | OpenEye OEToolkits 1.7.2 | |
|---|
-PDBエントリ
全3件を表示しています

PDB-1cvw: 
Crystal structure of active site-inhibited human coagulation factor VIIA (DES-GLA)

PDB-3th3: 
Mg2+ Is Required for Optimal Folding of the Gamma-Carboxyglutamic Acid (Gla) Domains of Vitamin K-Dependent Clotting Factors At Physiological Ca2+

PDB-3th4: 
Mg2+ Is Required for Optimal Folding of the Gamma-Carboxyglutamic Acid (Gla) Domains of Vitamin K-Dependent Clotting Factors At Physiological Ca2+
 ムービー
ムービー コントローラー
コントローラー