+検索条件
-Structure paper
| タイトル | Cryo-EM structure of ribosome from pathogenic protozoa Entamoeba histolytica reveals unique features of its architecture. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 16, Issue 1, Page 7758, Year 2025 |
| 掲載日 | 2025年8月20日 |
 著者 著者 | Shivani Sharma / Shalini Mishra / Samudrala Gourinath / Prem S Kaushal /  |
| PubMed 要旨 | Entamoeba histolytica, an anaerobic protozoan parasite, is the causative agent of amoebiasis, bloody diarrhea, and liver abscesses in humans. Amoebiasis is more predominant in tropical areas with ...Entamoeba histolytica, an anaerobic protozoan parasite, is the causative agent of amoebiasis, bloody diarrhea, and liver abscesses in humans. Amoebiasis is more predominant in tropical areas with poor sanitation conditions, and it remains the fourth leading cause of death due to a protozoan infection. E. histolytica life cycle spans between an infective 'cyst stage' and an active disease-causing 'trophozoite stage'. We have isolated ribosomes from the trophozoite stage of E. histolytica. Here, we report single particle cryo-EM structures of the 53S ribosome large subunit (LSU), and 75S associated ribosomes, with P-tRNA, A/P and P/E tRNAs, and with paromomycin antibiotic, at 2.8 Å to 3.4 Å resolution. The E. histolytica possesses a reduced ribosome with a conserved core, and the periphery evolved with species-specific unique features. The most notable features are the presence of the rRNA triple helix near the peptide exit tunnel, the expansion segment H88ES102 near the exit site on LSU, and unique insertions in r-proteins. Furthermore, the 75S ribosome paromomycin complex structure provides the atomic details of its interactions. These structures provide insights into the evolutionary adaptation of the E. histolytica translational machinery and may be explored further for amoebicidal therapeutic intervention. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:40835601 / PubMed:40835601 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.8 - 3.4 Å |
| 構造データ | EMDB-64694, PDB-9v1i: EMDB-64695, PDB-9v1j: EMDB-64696, PDB-9v1k: EMDB-64697, PDB-9v1l: EMDB-64713, PDB-9v21: EMDB-64716, PDB-9v24: EMDB-64717, PDB-9v25: EMDB-64718, PDB-9v26: EMDB-64719, PDB-9v27: EMDB-64720, PDB-9v28: EMDB-64721, PDB-9v29: |
| 化合物 | 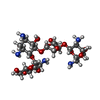 ChemComp-PAR: |
| 由来 |
|
 キーワード キーワード | RIBOSOME / LSU / Entamoeba histolytica / 53S / SSU-body / SSU-head / P-tRNA / LSU with P-tRNA CCA end / 75S Ribosome / A/P-tRNA / P/E-tRNA / Large Subunit of Ribosome / 75S Ribosome-PAR complex / SSU-body of 75S Ribosome / SSU-head of 75S Ribosome / SSU-body of 75S Ribosome-PAR complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について

























 entamoeba histolytica hm-1:imss (赤痢アメーバ)
entamoeba histolytica hm-1:imss (赤痢アメーバ)