+検索条件
-Structure paper
| タイトル | Structures of the nitrogenase complex prepared under catalytic turnover conditions. |
|---|---|
| ジャーナル・号・ページ | Science, Vol. 377, Issue 6608, Page 865-869, Year 2022 |
| 掲載日 | 2022年8月19日 |
 著者 著者 | Hannah L Rutledge / Brian D Cook / Hoang P M Nguyen / Mark A Herzik / F Akif Tezcan /  |
| PubMed 要旨 | The enzyme nitrogenase couples adenosine triphosphate (ATP) hydrolysis to the multielectron reduction of atmospheric dinitrogen into ammonia. Despite extensive research, the mechanistic details of ...The enzyme nitrogenase couples adenosine triphosphate (ATP) hydrolysis to the multielectron reduction of atmospheric dinitrogen into ammonia. Despite extensive research, the mechanistic details of ATP-dependent energy transduction and dinitrogen reduction by nitrogenase are not well understood, requiring new strategies to monitor its structural dynamics during catalytic action. Here, we report cryo-electron microscopy structures of the nitrogenase complex prepared under enzymatic turnover conditions. We observe that asymmetry governs all aspects of the nitrogenase mechanism, including ATP hydrolysis, protein-protein interactions, and catalysis. Conformational changes near the catalytic iron-molybdenum cofactor are correlated with the nucleotide-hydrolysis state of the enzyme. |
 リンク リンク |  Science / Science /  PubMed:35901182 / PubMed:35901182 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 1.91 - 3.01 Å |
| 構造データ | EMDB-26756, PDB-7ut6: EMDB-26757, PDB-7ut7:  EMDB-26758: Consensus cryoEM map of Azotobacter vinelandii MoFeP in a 1:1 complex (ATP-bound) with FeP during catalytic N2 reduction  EMDB-26759: Locally refined cryoEM map of Azotobacter vinelandii FeP in a 1:1 complex (ATP-bound) with MoFeP during catalytic N2 reduction EMDB-26760, PDB-7ut8:  EMDB-26761: Consensus cryoEM map of Azotobacter vinelandii MoFeP in a 1:1 complex (ADP/ATP-bound) with FeP during catalytic N2 reduction  EMDB-26762: Locally refined cryoEM map of Azotobacter vinelandii FeP in a 1:1 complex (ADP/ATP-bound) with MoFeP during catalytic N2 reduction EMDB-26763, PDB-7ut9: EMDB-26764, PDB-7uta: EMDB-27639, PDB-8dpn: |
| 化合物 | 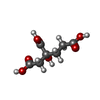 ChemComp-HCA: 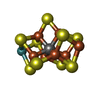 ChemComp-ICS:  ChemComp-FE: 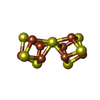 ChemComp-CLF:  ChemComp-HOH:  ChemComp-SF4:  ChemComp-MG:  ChemComp-ATP:  ChemComp-ADP:  ChemComp-0BE: |
| 由来 |
|
 キーワード キーワード | OXIDOREDUCTASE / nitrogenase / MoFeP / nitrogen fixation / FeP |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について















 azotobacter vinelandii dj (窒素固定)
azotobacter vinelandii dj (窒素固定)