+検索条件
-Structure paper
| タイトル | Viral DNA polymerase structures reveal mechanisms of antiviral drug resistance. |
|---|---|
| ジャーナル・号・ページ | Cell, Vol. 187, Issue 20, Page 5572-5586.e15, Year 2024 |
| 掲載日 | 2024年10月3日 |
 著者 著者 | Sundaresh Shankar / Junhua Pan / Pan Yang / Yuemin Bian / Gábor Oroszlán / Zishuo Yu / Purba Mukherjee / David J Filman / James M Hogle / Mrinal Shekhar / Donald M Coen / Jonathan Abraham /    |
| PubMed 要旨 | DNA polymerases are important drug targets, and many structural studies have captured them in distinct conformations. However, a detailed understanding of the impact of polymerase conformational ...DNA polymerases are important drug targets, and many structural studies have captured them in distinct conformations. However, a detailed understanding of the impact of polymerase conformational dynamics on drug resistance is lacking. We determined cryoelectron microscopy (cryo-EM) structures of DNA-bound herpes simplex virus polymerase holoenzyme in multiple conformations and interacting with antivirals in clinical use. These structures reveal how the catalytic subunit Pol and the processivity factor UL42 bind DNA to promote processive DNA synthesis. Unexpectedly, in the absence of an incoming nucleotide, we observed Pol in multiple conformations with the closed state sampled by the fingers domain. Drug-bound structures reveal how antivirals may selectively bind enzymes that more readily adopt the closed conformation. Molecular dynamics simulations and the cryo-EM structure of a drug-resistant mutant indicate that some resistance mutations modulate conformational dynamics rather than directly impacting drug binding, thus clarifying mechanisms that drive drug selectivity. |
 リンク リンク |  Cell / Cell /  PubMed:39197451 PubMed:39197451 |
| 手法 | EM (単粒子) |
| 解像度 | 2.7 - 5.1 Å |
| 構造データ | EMDB-28663, PDB-8exx:  EMDB-28664: Herpes simplex virus 1 DNA polymerase holoenzyme bound to DNA template and primer, dNTP-free (editing mode) EMDB-42887, PDB-8v1q: EMDB-42888, PDB-8v1r: EMDB-42889, PDB-8v1s: EMDB-42890, PDB-8v1t:  EMDB-42891: Herpes simplex virus 1 polymerase W781V mutant holoenzyme bound to DNA in editing conformation |
| 化合物 |  ChemComp-MG: 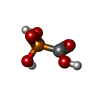 ChemComp-PPF:  ChemComp-HOH:  ChemComp-TTP: 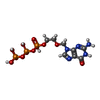 ChemComp-AVP: |
| 由来 |
|
 キーワード キーワード | VIRAL PROTEIN/DNA / herpes simplex virus / replication / DNA polymerase holoenzyme / UL30 / UL42 / pre-translocation / foscarnet / VIRAL PROTEIN / VIRAL PROTEIN-DNA complex / TRANSFERASE/DNA / open/closed conformations / conformational dynamics / TRANSFERASE-DNA complex / closed conformation / editing conformation / DNA polymerase / ACYCLOVIR / antiherpesvirus drug |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について














 Human herpesvirus 1 (strain KOS) (ヘルペスウイルス)
Human herpesvirus 1 (strain KOS) (ヘルペスウイルス)