+検索条件
-Structure paper
| タイトル | Molecular analysis and essentiality of Aro1 shikimate biosynthesis multi-enzyme in . |
|---|---|
| ジャーナル・号・ページ | Life Sci Alliance, Vol. 5, Issue 8, Year 2022 |
| 掲載日 | 2022年5月5日 |
 著者 著者 | Peter J Stogios / Sean D Liston / Cameron Semper / Bradley Quade / Karolina Michalska / Elena Evdokimova / Shane Ram / Zbyszek Otwinowski / Dominika Borek / Leah E Cowen / Alexei Savchenko /   |
| PubMed 要旨 | In the human fungal pathogen , encodes an essential multi-enzyme that catalyses consecutive steps in the shikimate pathway for biosynthesis of chorismate, a precursor to folate and the aromatic ...In the human fungal pathogen , encodes an essential multi-enzyme that catalyses consecutive steps in the shikimate pathway for biosynthesis of chorismate, a precursor to folate and the aromatic amino acids. We obtained the first molecular image of Aro1 that reveals the architecture of all five enzymatic domains and their arrangement in the context of the full-length protein. Aro1 forms a flexible dimer allowing relative autonomy of enzymatic function of the individual domains. Our activity and in cellulo data suggest that only four of Aro1's enzymatic domains are functional and essential for viability of , whereas the 3-dehydroquinate dehydratase (DHQase) domain is inactive because of active site substitutions. We further demonstrate that in , the type II DHQase Dqd1 can compensate for the inactive DHQase domain of Aro1, suggesting an unrecognized essential role for this enzyme in shikimate biosynthesis. In contrast, in and , which do not encode a Dqd1 homolog, Aro1 DHQase domains are enzymatically active, highlighting diversity across species. |
 リンク リンク |  Life Sci Alliance / Life Sci Alliance /  PubMed:35512834 / PubMed:35512834 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.85 - 4.16 Å |
| 構造データ | EMDB-26357, PDB-7u5s: EMDB-26358, PDB-7u5t: EMDB-26359, PDB-7u5u:  PDB-6c5c:  PDB-7tbu:  PDB-7tbv: |
| 化合物 |  ChemComp-NAD:  ChemComp-EDO:  ChemComp-CL:  ChemComp-HOH:  ChemComp-TRS: 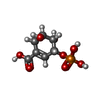 ChemComp-S3P:  ChemComp-MG:  ChemComp-GOL: |
| 由来 |
|
 キーワード キーワード | LYASE / chorismate biosynthesis / Structural Genomics / Center for Structural Genomics of Infectious Diseases / CSGID / TRANSFERASE / OXIDOREDUCTASE / BIOSYNTHETIC PROTEIN |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について









 candida albicans (酵母)
candida albicans (酵母)