+検索条件
-Structure paper
| タイトル | Structure of PDE3A-SLFN12 complex reveals requirements for activation of SLFN12 RNase. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 12, Issue 1, Page 4375, Year 2021 |
| 掲載日 | 2021年7月16日 |
 著者 著者 | Colin W Garvie / Xiaoyun Wu / Malvina Papanastasiou / Sooncheol Lee / James Fuller / Gavin R Schnitzler / Steven W Horner / Andrew Baker / Terry Zhang / James P Mullahoo / Lindsay Westlake / Stephanie H Hoyt / Marcus Toetzl / Matthew J Ranaghan / Luc de Waal / Joseph McGaunn / Bethany Kaplan / Federica Piccioni / Xiaoping Yang / Martin Lange / Adrian Tersteegen / Donald Raymond / Timothy A Lewis / Steven A Carr / Andrew D Cherniack / Christopher T Lemke / Matthew Meyerson / Heidi Greulich /   |
| PubMed 要旨 | DNMDP and related compounds, or velcrins, induce complex formation between the phosphodiesterase PDE3A and the SLFN12 protein, leading to a cytotoxic response in cancer cells that express elevated ...DNMDP and related compounds, or velcrins, induce complex formation between the phosphodiesterase PDE3A and the SLFN12 protein, leading to a cytotoxic response in cancer cells that express elevated levels of both proteins. The mechanisms by which velcrins induce complex formation, and how the PDE3A-SLFN12 complex causes cancer cell death, are not fully understood. Here, we show that PDE3A and SLFN12 form a heterotetramer stabilized by binding of DNMDP. Interactions between the C-terminal alpha helix of SLFN12 and residues near the active site of PDE3A are required for complex formation, and are further stabilized by interactions between SLFN12 and DNMDP. Moreover, we demonstrate that SLFN12 is an RNase, that PDE3A binding increases SLFN12 RNase activity, and that SLFN12 RNase activity is required for DNMDP response. This new mechanistic understanding will facilitate development of velcrin compounds into new cancer therapies. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:34272366 / PubMed:34272366 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.7 - 3.22 Å |
| 構造データ | EMDB-23494, PDB-7lrc: EMDB-23495, PDB-7lrd: EMDB-23496, PDB-7lre:  PDB-7kwe:  PDB-7l27:  PDB-7l28:  PDB-7l29: |
| 化合物 | 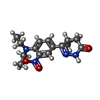 ChemComp-X5M:  ChemComp-MN:  ChemComp-MG:  ChemComp-ACT:  ChemComp-HOH:  ChemComp-CA: 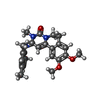 ChemComp-XKG:  ChemComp-AMP:  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード |  HYDROLASE (加水分解酵素) / HYDROLASE/HYDROLASE INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR complex / HYDROLASE (加水分解酵素) / HYDROLASE/HYDROLASE INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR complex /  complex / velcrin / complex / velcrin /  molecular glue / DNMDP / molecular glue / DNMDP /  RNA BINDING PROTEIN (RNA結合タンパク質) RNA BINDING PROTEIN (RNA結合タンパク質) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について










