+検索条件
-Structure paper
| タイトル | Synthetic group A streptogramin antibiotics that overcome Vat resistance. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 586, Issue 7827, Page 145-150, Year 2020 |
| 掲載日 | 2020年9月23日 |
 著者 著者 | Qi Li / Jenna Pellegrino / D John Lee / Arthur A Tran / Hector A Chaires / Ruoxi Wang / Jesslyn E Park / Kaijie Ji / David Chow / Na Zhang / Axel F Brilot / Justin T Biel / Gydo van Zundert / Kenneth Borrelli / Dean Shinabarger / Cindy Wolfe / Beverly Murray / Matthew P Jacobson / Estelle Mühle / Olivier Chesneau / James S Fraser / Ian B Seiple /    |
| PubMed 要旨 | Natural products serve as chemical blueprints for most antibiotics in clinical use. The evolutionary process by which these molecules arise is inherently accompanied by the co-evolution of resistance ...Natural products serve as chemical blueprints for most antibiotics in clinical use. The evolutionary process by which these molecules arise is inherently accompanied by the co-evolution of resistance mechanisms that shorten the clinical lifetime of any given class of antibiotics. Virginiamycin acetyltransferase (Vat) enzymes are resistance proteins that provide protection against streptogramins, potent antibiotics against Gram-positive bacteria that inhibit the bacterial ribosome. Owing to the challenge of selectively modifying the chemically complex, 23-membered macrocyclic scaffold of group A streptogramins, analogues that overcome the resistance conferred by Vat enzymes have not been previously developed. Here we report the design, synthesis, and antibacterial evaluation of group A streptogramin antibiotics with extensive structural variability. Using cryo-electron microscopy and forcefield-based refinement, we characterize the binding of eight analogues to the bacterial ribosome at high resolution, revealing binding interactions that extend into the peptidyl tRNA-binding site and towards synergistic binders that occupy the nascent peptide exit tunnel. One of these analogues has excellent activity against several streptogramin-resistant strains of Staphylococcus aureus, exhibits decreased rates of acetylation in vitro, and is effective at lowering bacterial load in a mouse model of infection. Our results demonstrate that the combination of rational design and modular chemical synthesis can revitalize classes of antibiotics that are limited by naturally arising resistance mechanisms. |
 リンク リンク |  Nature / Nature /  PubMed:32968273 / PubMed:32968273 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.5 - 3.05 Å |
| 構造データ | EMDB-20296, PDB-6pc5: EMDB-20297, PDB-6pc6: EMDB-20298, PDB-6pc7: EMDB-20299, PDB-6pc8: EMDB-20300, PDB-6pch: EMDB-20304, PDB-6pcq: EMDB-20305, PDB-6pcr: EMDB-20306, PDB-6pcs: EMDB-20307, PDB-6pct: EMDB-21969, PDB-6wyv:  PDB-6x3c:  PDB-6x3j: |
| 化合物 |  ChemComp-O7V: 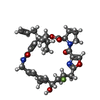 ChemComp-O7S:  ChemComp-O7Y: 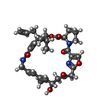 ChemComp-O8D:  ChemComp-O8J:  ChemComp-O8P:  ChemComp-O8S:  ChemComp-O8V:  ChemComp-SXA:  ChemComp-PO4:  ChemComp-SO4:  ChemComp-CL:  ChemComp-MG:  ChemComp-HOH: |
| 由来 |
|
 キーワード キーワード | RIBOSOME / E. coli ribosome / streptogramin A analog / antibiotics / TRANSFERASE / Acetyltransferase |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について
























