+検索条件
-Structure paper
| タイトル | Structural basis of norepinephrine recognition and transport inhibition in neurotransmitter transporters. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 12, Page 2199-2199, Year 2021 |
| 掲載日 | 2020年2月21日 (構造データの登録日) |
 著者 著者 | Pidathala, S. / Mallela, A.K. / Joseph, D. / Penmatsa, A. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:33850134 PubMed:33850134 |
| 手法 | X線回折 |
| 解像度 | 2.802 - 3.3 Å |
| 構造データ |  PDB-6m0f:  PDB-6m0z:  PDB-6m2r:  PDB-6m38:  PDB-6m3z:  PDB-6m47: |
| 化合物 |  ChemComp-CL:  ChemComp-CLR:  ChemComp-P4G:  ChemComp-NA:  ChemComp-HOH:  ChemComp-Y01: 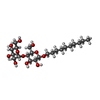 ChemComp-DMU: 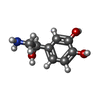 ChemComp-LNR:  ChemComp-D10: 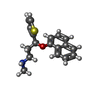 ChemComp-29E: 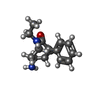 ChemComp-F0F:  ChemComp-GOL:  ChemComp-F1U: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / neurotransmitter transporter / antibody fragment |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





