+検索条件
-Structure paper
| タイトル | Structural basis for perception of diverse chemical substances by T1r taste receptors |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 8, Page 15530-15530, Year 2017 |
| 掲載日 | 2017年2月2日 (構造データの登録日) |
 著者 著者 | Nuemket, N. / Yasui, N. / Kusakabe, Y. / Nomura, Y. / Atsumi, N. / Akiyama, S. / Nango, E. / Kato, Y. / Kaneko, M.K. / Takagi, J. ...Nuemket, N. / Yasui, N. / Kusakabe, Y. / Nomura, Y. / Atsumi, N. / Akiyama, S. / Nango, E. / Kato, Y. / Kaneko, M.K. / Takagi, J. / Hosotani, M. / Yamashita, A. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:28534491 PubMed:28534491 |
| 手法 | X線回折 |
| 解像度 | 2.2 - 2.608 Å |
| 構造データ |  PDB-5x2m:  PDB-5x2n:  PDB-5x2o:  PDB-5x2p:  PDB-5x2q: |
| 化合物 |  ChemComp-NAG: 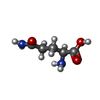 ChemComp-GLN:  ChemComp-NA:  ChemComp-CL:  ChemComp-CA:  ChemComp-HOH: 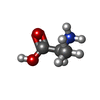 ChemComp-ALA:  ChemComp-ARG:  ChemComp-GLU:  ChemComp-GLY: |
| 由来 |
|
 キーワード キーワード | SIGNALING PROTEIN/IMMUNE SYSTEM / receptor / ligand binding / amino acid / venus-flytrap domain / SIGNALING PROTEIN-IMMUNE SYSTEM complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





