+検索条件
-Structure paper
| タイトル | Structural Analysis of Glycine Sarcosine N-methyltransferase from Methanohalophilus portucalensis Reveals Mechanistic Insights into the Regulation of Methyltransferase Activity. |
|---|---|
| ジャーナル・号・ページ | Sci Rep, Vol. 6, Page 38071-38071, Year 2016 |
| 掲載日 | 2016年9月14日 (構造データの登録日) |
 著者 著者 | Lee, Y.R. / Lin, T.S. / Lai, S.J. / Liu, M.S. / Lai, M.C. / Chan, N.L. |
 リンク リンク |  Sci Rep / Sci Rep /  PubMed:27934872 PubMed:27934872 |
| 手法 | X線回折 |
| 解像度 | 1.776 - 2.471 Å |
| 構造データ |  PDB-5gwx:  PDB-5h02:  PDB-5hii:  PDB-5hij:  PDB-5hik:  PDB-5hil:  PDB-5him: |
| 化合物 |  ChemComp-SAM:  ChemComp-SAR:  ChemComp-EDO:  ChemComp-HOH:  ChemComp-SAH:  ChemComp-BET: 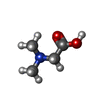 ChemComp-DMG: |
| 由来 |
|
 キーワード キーワード | TRANSFERASE / S-adenosylmethionine-dependent methyltransferases / SAM / AdoMet-MTase / class I monomethylation / glycine / sarcosine rate-limiting enzyme / betaine biosynthesis betaine-mediated feedback inhibition / sarcosine / rate-limiting enzyme / betaine biosynthesis / betaine-mediated feedback inhibition / S-adenosylmethionine-dependent methyltransferases (SAM or AdoMet-MTase) / class I / monomethylation of glycine and sarcosine / rate-limiting enzyme in betaine biosynthesis |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 methanohalophilus portucalensis fdf-1 (古細菌)
methanohalophilus portucalensis fdf-1 (古細菌)