+検索条件
-Structure paper
| タイトル | A Maltose-Binding Protein Fusion Construct Yields a Robust Crystallography Platform for MCL1. |
|---|---|
| ジャーナル・号・ページ | Plos One, Vol. 10, Page e0125010-e0125010, Year 2015 |
| 掲載日 | 2014年10月9日 (構造データの登録日) |
 著者 著者 | Clifton, M.C. / Dranow, D.M. / Leed, A. / Fulroth, B. / Fairman, J.W. / Abendroth, J. / Atkins, K.A. / Wallace, E. / Fan, D. / Xu, G. ...Clifton, M.C. / Dranow, D.M. / Leed, A. / Fulroth, B. / Fairman, J.W. / Abendroth, J. / Atkins, K.A. / Wallace, E. / Fan, D. / Xu, G. / Ni, Z.J. / Daniels, D. / Van Drie, J. / Wei, G. / Burgin, A.B. / Golub, T.R. / Hubbard, B.K. / Serrano-Wu, M.H. |
 リンク リンク |  Plos One / Plos One /  PubMed:25909780 PubMed:25909780 |
| 手法 | X線回折 |
| 解像度 | 1.55 - 2.4 Å |
| 構造データ |  PDB-4wmr:  PDB-4wms:  PDB-4wmt:  PDB-4wmu:  PDB-4wmv:  PDB-4wmw:  PDB-4wmx: |
| 化合物 |  ChemComp-ZN: 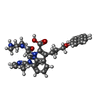 ChemComp-865:  ChemComp-POP:  ChemComp-HOH:  ChemComp-MG:  ChemComp-EDO:  ChemComp-FMT: 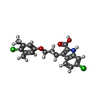 ChemComp-19H:  ChemComp-NA:  ChemComp-CL: 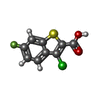 ChemComp-3R4:  ChemComp-3R6: 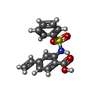 ChemComp-3R7: |
| 由来 |
|
 キーワード キーワード | Apoptosis/Inhibitor / Apoptosis / protein-protein interaction / Apoptosis-Inhibitor complex / chimera protein |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)
