+検索条件
-Structure paper
| タイトル | Crystal structures of the effector-binding domain of repressor Central glycolytic gene Regulator from Bacillus subtilis reveal ligand-induced structural changes upon binding of several glycolytic intermediates. |
|---|---|
| ジャーナル・号・ページ | Mol. Microbiol., Vol. 69, Page 895-910, Year 2008 |
| 掲載日 | 2007年1月16日 (構造データの登録日) |
 著者 著者 | Rezacova, P. / Kozisek, M. / Moy, S.F. / Sieglova, I. / Joachimiak, A. / Machius, M. / Otwinowski, Z. |
 リンク リンク |  Mol. Microbiol. / Mol. Microbiol. /  PubMed:18554327 PubMed:18554327 |
| 手法 | X線回折 |
| 解像度 | 1.65 - 1.85 Å |
| 構造データ |  PDB-2okg:  PDB-3bxe:  PDB-3bxf:  PDB-3bxg:  PDB-3bxh: |
| 化合物 |  ChemComp-CL: 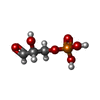 ChemComp-G3H:  ChemComp-HOH: 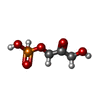 ChemComp-13P: 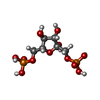 ChemComp-FBP: 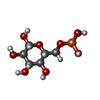 ChemComp-BG6:  ChemComp-F6P:  ChemComp-SCN: |
| 由来 |
|
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / alpha/beta/alpha sandwich / Rossmann-like fold / TRANSCRIPTION (転写 (生物学)) / alpha/beta/alpha sandwich / Rossmann-like fold /  Structural Genomics (構造ゲノミクス) / PSI-2 / Structural Genomics (構造ゲノミクス) / PSI-2 /  Protein Structure Initiative / Midwest Center for Structural Genomics / MCSG / Protein Structure Initiative / Midwest Center for Structural Genomics / MCSG /  GENE REGULATION (遺伝子発現の調節) / effector binding domain / catabolic repressor / GENE REGULATION (遺伝子発現の調節) / effector binding domain / catabolic repressor /  transcriptional regulator / transcriptional regulator /  DeoR family / DNA-binding / DeoR family / DNA-binding /  Transcription regulation Transcription regulation |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




