+検索条件
-Structure paper
| タイトル | Structural Basis for Lower Lysine Methylation State-Specific Readout by MBT Repeats of L3MBTL1 and an Engineered PHD Finger. |
|---|---|
| ジャーナル・号・ページ | Mol. Cell, Vol. 28, Page 677-691, Year 2007 |
| 掲載日 | 2007年10月9日 (構造データの登録日) |
 著者 著者 | Li, H. / Fischle, W. / Wang, W. / Duncan, E.M. / Liang, L. / Murakami-Ishibe, S. / Allis, C.D. / Patel, D.J. |
 リンク リンク |  Mol. Cell / Mol. Cell /  PubMed:18042461 PubMed:18042461 |
| 手法 | X線回折 |
| 解像度 | 1.45 - 2.2 Å |
| 構造データ |  PDB-2rhi:  PDB-2rhu:  PDB-2rhx:  PDB-2rhy:  PDB-2rhz:  PDB-2ri2:  PDB-2ri3:  PDB-2ri5: 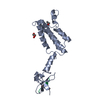 PDB-2ri7: |
| 化合物 |  ChemComp-PG4:  ChemComp-PEG:  ChemComp-HOH:  ChemComp-SO4: 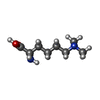 ChemComp-MLY:  ChemComp-PGE: 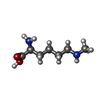 ChemComp-MLZ:  ChemComp-ZN:  ChemComp-GOL:  ChemComp-IPA: |
| 由来 |
|
 キーワード キーワード | Transcription/Nuclear Protein / BETA BARREL / Protein-peptide Complex / dimethyl-lysine / Alternative splicing / Chromatin regulator / DNA-binding / Metal-binding / Nucleus / Repressor / Transcription / Transcription regulation / Zinc / Zinc-finger / Transcription-Nuclear Protein COMPLEX / monomethyl-lysine / Zinc finger / alpha-helical bundle / Bromodomain / Phosphorylation |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)