+検索条件
-Structure paper
| タイトル | Ligand binding and conformational changes of SUR1 subunit in pancreatic ATP-sensitive potassium channels. |
|---|---|
| ジャーナル・号・ページ | Protein Cell, Vol. 9, Issue 6, Page 553-567, Year 2018 |
| 掲載日 | 2018年3月28日 |
 著者 著者 | Jing-Xiang Wu / Dian Ding / Mengmeng Wang / Yunlu Kang / Xin Zeng / Lei Chen /  |
| PubMed 要旨 | ATP-sensitive potassium channels (K) are energy sensors on the plasma membrane. By sensing the intracellular ADP/ATP ratio of β-cells, pancreatic K channels control insulin release and regulate ...ATP-sensitive potassium channels (K) are energy sensors on the plasma membrane. By sensing the intracellular ADP/ATP ratio of β-cells, pancreatic K channels control insulin release and regulate metabolism at the whole body level. They are implicated in many metabolic disorders and diseases and are therefore important drug targets. Here, we present three structures of pancreatic K channels solved by cryo-electron microscopy (cryo-EM), at resolutions ranging from 4.1 to 4.5 Å. These structures depict the binding site of the antidiabetic drug glibenclamide, indicate how Kir6.2 (inward-rectifying potassium channel 6.2) N-terminus participates in the coupling between the peripheral SUR1 (sulfonylurea receptor 1) subunit and the central Kir6.2 channel, reveal the binding mode of activating nucleotides, and suggest the mechanism of how Mg-ADP binding on nucleotide binding domains (NBDs) drives a conformational change of the SUR1 subunit. |
 リンク リンク |  Protein Cell / Protein Cell /  PubMed:29594720 / PubMed:29594720 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 4.11 - 6.1 Å |
| 構造データ | EMDB-6831, PDB-5yke: EMDB-6832, PDB-5ykf: EMDB-6833, PDB-5ykg: EMDB-6847, PDB-5yw7: EMDB-6848, PDB-5yw8: EMDB-6849, PDB-5yw9: EMDB-6850, PDB-5ywa: EMDB-6851, PDB-5ywb: |
| 化合物 | 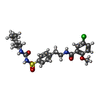 ChemComp-GBM:  ChemComp-AGS:  ChemComp-ADP:  ChemComp-MG: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / KATP / channel / glibenclamide / sulfonylurea |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について
























