+検索条件
-Structure paper
| タイトル | Probing the mechanism by which the retinal G protein transducin activates its biological effector PDE6. |
|---|---|
| ジャーナル・号・ページ | J Biol Chem, Vol. 300, Issue 2, Page 105608, Year 2024 |
| 掲載日 | 2023年12月28日 |
 著者 著者 | Cody Aplin / Richard A Cerione /  |
| PubMed 要旨 | Phototransduction in retinal rods occurs when the G protein-coupled photoreceptor rhodopsin triggers the activation of phosphodiesterase 6 (PDE6) by GTP-bound alpha subunits of the G protein ...Phototransduction in retinal rods occurs when the G protein-coupled photoreceptor rhodopsin triggers the activation of phosphodiesterase 6 (PDE6) by GTP-bound alpha subunits of the G protein transducin (Gα). Recently, we presented a cryo-EM structure for a complex between two GTP-bound recombinant Gα subunits and native PDE6, that included a bivalent antibody bound to the C-terminal ends of Gα and the inhibitor vardenafil occupying the active sites on the PDEα and PDEβ subunits. We proposed Gα-activated PDE6 by inducing a striking reorientation of the PDEγ subunits away from the catalytic sites. However, questions remained including whether in the absence of the antibody Gα binds to PDE6 in a similar manner as observed when the antibody is present, does Gα activate PDE6 by enabling the substrate cGMP to access the catalytic sites, and how does the lipid membrane enhance PDE6 activation? Here, we demonstrate that 2:1 Gα-PDE6 complexes form with either recombinant or retinal Gα in the absence of the Gα antibody. We show that Gα binding is not necessary for cGMP nor competitive inhibitors to access the active sites; instead, occupancy of the substrate binding sites enables Gα to bind and reposition the PDE6γ subunits to promote catalytic activity. Moreover, we demonstrate by reconstituting Gα-stimulated PDE6 activity in lipid bilayer nanodiscs that the membrane-induced enhancement results from an increase in the apparent binding affinity of Gα for PDE6. These findings provide new insights into how the retinal G protein stimulates rapid catalytic turnover by PDE6 required for dim light vision. |
 リンク リンク |  J Biol Chem / J Biol Chem /  PubMed:38159849 / PubMed:38159849 /  PubMed Central PubMed Central |
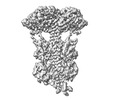
| 手法 | EM (単粒子) |
| 解像度 | 3.0 - 4.44 Å |
| 構造データ | EMDB-42208, PDB-8ufi: EMDB-42220, PDB-8ugb: EMDB-42234, PDB-8ugs:  EMDB-42235: Bovine rod phosphodiesterase 6 bound to one retinal transducin alpha subunit  EMDB-42237: Bovine rod phosphodiesterase 6 bound to two retinal transducin alpha subunits  EMDB-42238: Bovine rod phosphodiesterase 6 bound to two chimera transducin alpha subunits without a stabilizing antibody EMDB-42358, PDB-8ulg: |
| 化合物 |  ChemComp-PCG:  ChemComp-ZN:  ChemComp-MG: 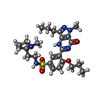 ChemComp-ZUD:  ChemComp-IBM: |
| 由来 |
|
 キーワード キーワード | SIGNALING PROTEIN / phosphodiesterase / GPCR effector enzyme / SIGNALING PROTEIN/INHIBITOR / inhibitor / SIGNALING PROTEIN-INHIBITOR complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について