+検索条件
-Structure paper
| タイトル | Structural basis for urate recognition and apigenin inhibition of human GLUT9. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 15, Issue 1, Page 5039, Year 2024 |
| 掲載日 | 2024年6月12日 |
 著者 著者 | Zilin Shen / Li Xu / Tong Wu / Huan Wang / Qifan Wang / Xiaofei Ge / Fang Kong / Gaoxingyu Huang / Xiaojing Pan /  |
| PubMed 要旨 | Urate, the physiological form of uric acid and a potent antioxidant in serum, plays a pivotal role in scavenging reactive oxygen species. Yet excessive accumulation of urate, known as hyperuricemia, ...Urate, the physiological form of uric acid and a potent antioxidant in serum, plays a pivotal role in scavenging reactive oxygen species. Yet excessive accumulation of urate, known as hyperuricemia, is the primary risk factor for the development of gout. The high-capacity urate transporter GLUT9 represents a promising target for gout treatment. Here, we present cryo-electron microscopy structures of human GLUT9 in complex with urate or its inhibitor apigenin at overall resolutions of 3.5 Å and 3.3 Å, respectively. In both structures, GLUT9 exhibits an inward open conformation, wherein the substrate binding pocket faces the intracellular side. These structures unveil the molecular basis for GLUT9's substrate preference of urate over glucose, and show that apigenin acts as a competitive inhibitor by occupying the substrate binding site. Our findings provide critical information for the development of specific inhibitors targeting GLUT9 as potential therapeutics for gout and hyperuricemia. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:38866775 / PubMed:38866775 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.28 - 3.51 Å |
| 構造データ | EMDB-38966, PDB-8y65: EMDB-38968, PDB-8y66: |
| 化合物 |  ChemComp-URC: 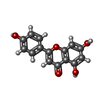 ChemComp-AGI: |
| 由来 |
|
 キーワード キーワード | TRANSPORT PROTEIN / GLUT9 / Urate / inhibitor / apigenin |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について







 homo sapiens (ヒト)
homo sapiens (ヒト)