+検索条件
-Structure paper
| タイトル | Structure of a SIN3-HDAC complex from budding yeast. |
|---|---|
| ジャーナル・号・ページ | Nat Struct Mol Biol, Vol. 30, Issue 6, Page 753-760, Year 2023 |
| 掲載日 | 2023年4月20日 |
 著者 著者 | Zhouyan Guo / Chen Chu / Yichen Lu / Xiaofeng Zhang / Yihang Xiao / Mingxuan Wu / Shuaixin Gao / Catherine C L Wong / Xiechao Zhan / Chengcheng Wang /   |
| PubMed 要旨 | SIN3-HDAC (histone deacetylases) complexes have important roles in facilitating local histone deacetylation to regulate chromatin accessibility and gene expression. Here, we present the cryo-EM ...SIN3-HDAC (histone deacetylases) complexes have important roles in facilitating local histone deacetylation to regulate chromatin accessibility and gene expression. Here, we present the cryo-EM structure of the budding yeast SIN3-HDAC complex Rpd3L at an average resolution of 2.6 Å. The structure reveals that two distinct arms (ARM1 and ARM2) hang on a T-shaped scaffold formed by two coiled-coil domains. In each arm, Sin3 interacts with different subunits to create a different environment for the histone deacetylase Rpd3. ARM1 is in the inhibited state with the active site of Rpd3 blocked, whereas ARM2 is in an open conformation with the active site of Rpd3 exposed to the exterior space. The observed asymmetric architecture of Rpd3L is different from those of available structures of other class I HDAC complexes. Our study reveals the organization mechanism of the SIN3-HDAC complex and provides insights into the interaction pattern by which it targets histone deacetylase to chromatin. |
 リンク リンク |  Nat Struct Mol Biol / Nat Struct Mol Biol /  PubMed:37081318 PubMed:37081318 |
| 手法 | EM (単粒子) |
| 解像度 | 2.6 - 4.5 Å |
| 構造データ | EMDB-34935, PDB-8hpo: 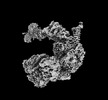 EMDB-34936: Cryo-EM map of the Rpd3L complex from the Rxt1-Flag dataset  EMDB-34937: Cryo-EM map of the Rpd3L complex in Sin3 part  EMDB-34938: Cryo-EM map of the Rpd3L complex in Dep1 region 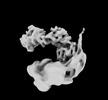 EMDB-34939: Cryo-EM map of the Rpd3L complex in Ume1 part |
| 化合物 | 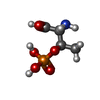 ChemComp-TPO:  ChemComp-ZN:  ChemComp-K: |
| 由来 |
|
 キーワード キーワード | DNA BINDING PROTEIN / SIN3 / HDAC / deacetylase / Rpd3 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について






