+検索条件
-Structure paper
| タイトル | Rcf2 revealed in cryo-EM structures of hypoxic isoforms of mature mitochondrial III-IV supercomplexes. |
|---|---|
| ジャーナル・号・ページ | Proc Natl Acad Sci U S A, Vol. 117, Issue 17, Page 9329-9337, Year 2020 |
| 掲載日 | 2020年4月28日 |
 著者 著者 | Andrew M Hartley / Brigitte Meunier / Nikos Pinotsis / Amandine Maréchal /   |
| PubMed 要旨 | The organization of the mitochondrial electron transport chain proteins into supercomplexes (SCs) is now undisputed; however, their assembly process, or the role of differential expression isoforms, ...The organization of the mitochondrial electron transport chain proteins into supercomplexes (SCs) is now undisputed; however, their assembly process, or the role of differential expression isoforms, remain to be determined. In , cytochrome oxidase (CIV) forms SCs of varying stoichiometry with cytochrome (CIII). Recent studies have revealed, in normoxic growth conditions, an interface made exclusively by Cox5A, the only yeast respiratory protein that exists as one of two isoforms depending on oxygen levels. Here we present the cryo-EM structures of the III-IV and III-IV SCs containing the hypoxic isoform Cox5B solved at 3.4 and 2.8 Å, respectively. We show that the change of isoform does not affect SC formation or activity, and that SC stoichiometry is dictated by the level of CIII/CIV biosynthesis. Comparison of the CIV- and CIV-containing SC structures highlighted few differences, found mainly in the region of Cox5. Additional density was revealed in all SCs, independent of the CIV isoform, in a pocket formed by Cox1, Cox3, Cox12, and Cox13, away from the CIII-CIV interface. In the CIV-containing hypoxic SCs, this could be confidently assigned to the hypoxia-induced gene 1 (Hig1) type 2 protein Rcf2. With conserved residues in mammalian Hig1 proteins and Cox3/Cox12/Cox13 orthologs, we propose that Hig1 type 2 proteins are stoichiometric subunits of CIV, at least when within a III-IV SC. |
 リンク リンク |  Proc Natl Acad Sci U S A / Proc Natl Acad Sci U S A /  PubMed:32291341 / PubMed:32291341 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.8 - 3.41 Å |
| 構造データ |  EMDB-10317: EMDB-10318: Complex IV of the III2-IV5B1 respiratory supercomplex from S. cerevisiae after focused refinement  EMDB-10334:  EMDB-10335: EMDB-10340, PDB-6t0b:  EMDB-10375:  EMDB-10376: |
| 化合物 |  ChemComp-CDL:  ChemComp-PEF:  ChemComp-HEM: 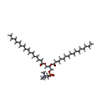 ChemComp-PCF:  ChemComp-HEC:  ChemComp-FES:  ChemComp-CU:  ChemComp-HEA:  ChemComp-CA:  ChemComp-MG:  ChemComp-CUA:  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード | OXIDOREDUCTASE / CYTOCHROME C OXIDASE CYTOCHROME BC1 MITOCHONDRIA RESPIRATORY CHAIN SUPERCOMPLEX / ELECTRON TRANSPORT / OXIDOREDUCTASE-ELECTRON TRANSPORT COMPLEX |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について








