+検索条件
-Structure paper
| タイトル | Inhibition of the Staphylococcus aureus c-di-AMP cyclase DacA by direct interaction with the phosphoglucosamine mutase GlmM. |
|---|---|
| ジャーナル・号・ページ | PLoS Pathog, Vol. 15, Issue 1, Page e1007537, Year 2019 |
| 掲載日 | 2019年1月22日 |
 著者 著者 | Tommaso Tosi / Fumiya Hoshiga / Charlotte Millership / Rahul Singh / Charles Eldrid / Delphine Patin / Dominique Mengin-Lecreulx / Konstantinos Thalassinos / Paul Freemont / Angelika Gründling /   |
| PubMed 要旨 | c-di-AMP is an important second messenger molecule that plays a pivotal role in regulating fundamental cellular processes, including osmotic and cell wall homeostasis in many Gram-positive organisms. ...c-di-AMP is an important second messenger molecule that plays a pivotal role in regulating fundamental cellular processes, including osmotic and cell wall homeostasis in many Gram-positive organisms. In the opportunistic human pathogen Staphylococcus aureus, c-di-AMP is produced by the membrane-anchored DacA enzyme. Inactivation of this enzyme leads to a growth arrest under standard laboratory growth conditions and a re-sensitization of methicillin-resistant S. aureus (MRSA) strains to ß-lactam antibiotics. The gene coding for DacA is part of the conserved three-gene dacA/ybbR/glmM operon that also encodes the proposed DacA regulator YbbR and the essential phosphoglucosamine mutase GlmM, which is required for the production of glucosamine-1-phosphate, an early intermediate of peptidoglycan synthesis. These three proteins are thought to form a complex in vivo and, in this manner, help to fine-tune the cellular c-di-AMP levels. To further characterize this important regulatory complex, we conducted a comprehensive structural and functional analysis of the S. aureus DacA and GlmM enzymes by determining the structures of the S. aureus GlmM enzyme and the catalytic domain of DacA. Both proteins were found to be dimers in solution as well as in the crystal structures. Further site-directed mutagenesis, structural and enzymatic studies showed that multiple DacA dimers need to interact for enzymatic activity. We also show that DacA and GlmM form a stable complex in vitro and that S. aureus GlmM, but not Escherichia coli or Pseudomonas aeruginosa GlmM, acts as a strong inhibitor of DacA function without the requirement of any additional cellular factor. Based on Small Angle X-ray Scattering (SAXS) data, a model of the complex revealed that GlmM likely inhibits DacA by masking the active site of the cyclase and preventing higher oligomer formation. Together these results provide an important mechanistic insight into how c-di-AMP production can be regulated in the cell. |
 リンク リンク |  PLoS Pathog / PLoS Pathog /  PubMed:30668586 / PubMed:30668586 /  PubMed Central PubMed Central |
| 手法 | SAS (X-ray synchrotron) / X線回折 |
| 解像度 | 1.7 - 3 Å |
| 構造データ |  SASDE78:  SASDE88:  SASDE98:  PDB-6gyw:  PDB-6gyx:  PDB-6gyy:  PDB-6gyz: |
| 化合物 |  ChemComp-HOH: 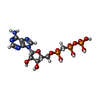 ChemComp-APC:  ChemComp-MN: |
| 由来 |
|
 キーワード キーワード | TRANSFERASE / c-di-AMP / diadenylate cyclase / ApCpp / protein engineering / ISOMERASE / cell wall / phosphoglucosamine mutase / peptidoglycan |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 Staphylococcus aureus (strain usa300) (黄色ブドウ球菌)
Staphylococcus aureus (strain usa300) (黄色ブドウ球菌)