+検索条件
-Structure paper
| タイトル | Structural and thermodynamic correlation illuminates the selective transport mechanism of disaccharide alpha-glycosides through ABC transporter. |
|---|---|
| ジャーナル・号・ページ | Febs J., Vol. 287, Page 1576-1597, Year 2020 |
| 掲載日 | 2019年1月24日 (構造データの登録日) |
 著者 著者 | Chandravanshi, M. / Gogoi, P. / Kanaujia, S.P. |
 リンク リンク |  Febs J. / Febs J. /  PubMed:31608555 PubMed:31608555 |
| 手法 | X線回折 |
| 解像度 | 1.56 - 2.1 Å |
| 構造データ |  PDB-6j9w:  PDB-6j9y:  PDB-6jad:  PDB-6jag:  PDB-6jah:  PDB-6jai:  PDB-6jal:  PDB-6jam:  PDB-6jan:  PDB-6jao:  PDB-6jap:  PDB-6jaq:  PDB-6jar:  PDB-6jaz:  PDB-6jb0:  PDB-6jb4:  PDB-6jba:  PDB-6jbb:  PDB-6jbe: |
| 化合物 |  ChemComp-CIT:  ChemComp-EDO:  ChemComp-HOH:  ChemComp-CO3:  ChemComp-GOL:  ChemComp-GLC:  ChemComp-PEG: 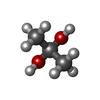 ChemComp-BA9: 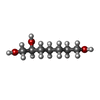 ChemComp-BA6:  ChemComp-CA:  ChemComp-ACT: 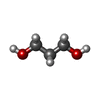 ChemComp-PDO: 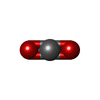 ChemComp-CO2: |
| 由来 |
|
 キーワード キーワード | SUGAR BINDING PROTEIN / Carbohydrate-binding site / alpha-glycoside-binding protein / Ligand selection / Multi-substrate transporter /  Sugar replacement / Venus Fly-trap mechanism / Carbohydrate-bindingsite Sugar replacement / Venus Fly-trap mechanism / Carbohydrate-bindingsite |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





