+検索条件
-Structure paper
| タイトル | N-terminal domain antigenic mapping reveals a site of vulnerability for SARS-CoV-2. |
|---|---|
| ジャーナル・号・ページ | Cell, Vol. 184, Issue 9, Page 2332-2347.e16, Year 2021 |
| 掲載日 | 2021年4月29日 |
 著者 著者 | Matthew McCallum / Anna De Marco / Florian A Lempp / M Alejandra Tortorici / Dora Pinto / Alexandra C Walls / Martina Beltramello / Alex Chen / Zhuoming Liu / Fabrizia Zatta / Samantha Zepeda / Julia di Iulio / John E Bowen / Martin Montiel-Ruiz / Jiayi Zhou / Laura E Rosen / Siro Bianchi / Barbara Guarino / Chiara Silacci Fregni / Rana Abdelnabi / Shi-Yan Caroline Foo / Paul W Rothlauf / Louis-Marie Bloyet / Fabio Benigni / Elisabetta Cameroni / Johan Neyts / Agostino Riva / Gyorgy Snell / Amalio Telenti / Sean P J Whelan / Herbert W Virgin / Davide Corti / Matteo Samuele Pizzuto / David Veesler /      |
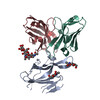
| PubMed 要旨 | The SARS-CoV-2 spike (S) glycoprotein contains an immunodominant receptor-binding domain (RBD) targeted by most neutralizing antibodies (Abs) in COVID-19 patient plasma. Little is known about ...The SARS-CoV-2 spike (S) glycoprotein contains an immunodominant receptor-binding domain (RBD) targeted by most neutralizing antibodies (Abs) in COVID-19 patient plasma. Little is known about neutralizing Abs binding to epitopes outside the RBD and their contribution to protection. Here, we describe 41 human monoclonal Abs (mAbs) derived from memory B cells, which recognize the SARS-CoV-2 S N-terminal domain (NTD) and show that a subset of them neutralize SARS-CoV-2 ultrapotently. We define an antigenic map of the SARS-CoV-2 NTD and identify a supersite (designated site i) recognized by all known NTD-specific neutralizing mAbs. These mAbs inhibit cell-to-cell fusion, activate effector functions, and protect Syrian hamsters from SARS-CoV-2 challenge, albeit selecting escape mutants in some animals. Indeed, several SARS-CoV-2 variants, including the B.1.1.7, B.1.351, and P.1 lineages, harbor frequent mutations within the NTD supersite, suggesting ongoing selective pressure and the importance of NTD-specific neutralizing mAbs for protective immunity and vaccine design. |
 リンク リンク |  Cell / Cell /  PubMed:33761326 / PubMed:33761326 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.2 - 6.5 Å |
| 構造データ | EMDB-23577, PDB-7lxw: EMDB-23578, PDB-7lxx: EMDB-23579, PDB-7lxy: EMDB-23580, PDB-7lxz: EMDB-23581, PDB-7ly0: EMDB-23582, PDB-7ly2:  EMDB-23583:  EMDB-23584:  EMDB-23585:  EMDB-23586:  PDB-7ly3: |
| 化合物 |  ChemComp-NAG:  ChemComp-HOH:  ChemComp-UNX:  ChemComp-XYL:  ChemComp-SO4: |
| 由来 |
|
 キーワード キーワード | VIRAL PROTEIN/IMMUNE SYSTEM / Antibody / VIRAL PROTEIN / Structural Genomics / Structural Genomics Consortium / SGC / Seattle Structural Genomics Center for Infectious Disease / SSGCID / VIRAL PROTEIN-IMMUNE SYSTEM complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN文献について
EMN文献について














 homo sapiens (ヒト)
homo sapiens (ヒト)

