| タイトル | Exploring Possible Drug-Resistant Variants of SARS-CoV-2 Main Protease (M pro ) with Noncovalent Preclinical Candidate, Mpro61. |
|---|
| ジャーナル・号・ページ | Acs Bio Med Chem Au, Vol. 5, Page 215-226, Year 2025 |
|---|
| 掲載日 | 2024年5月3日 (構造データの登録日) |
|---|
 著者 著者 | Kenneson, J.R. / Papini, C. / Tang, S. / Huynh, K. / Zhang, C.H. / Jorgensen, W.L. / Anderson, K.S. |
|---|
 リンク リンク |  Acs Bio Med Chem Au / Acs Bio Med Chem Au /  PubMed:39990941 PubMed:39990941 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.3 - 2.78 Å |
|---|
| 構造データ | PDB-9bnt:
Crystal Structure of T21I SARS-CoV-2 Main Protease
手法: X-RAY DIFFRACTION / 解像度: 1.83 Å PDB-9bnu:
Crystal Structure of T190I SARS-CoV-2 Main Protease
手法: X-RAY DIFFRACTION / 解像度: 1.55 Å PDB-9bnv:
Crystal Structure of A173V SARS-CoV-2 Main Protease
手法: X-RAY DIFFRACTION / 解像度: 1.67 Å PDB-9bnw:
Crystal Structure of A173V/L50F SARS-CoV-2 Main Protease
手法: X-RAY DIFFRACTION / 解像度: 1.3 Å PDB-9bnx:
Crystal Structure of L50F SARS-CoV-2 Main Protease
手法: X-RAY DIFFRACTION / 解像度: 2.48 Å PDB-9bny:
Crystal Structure of E166V/L50F SARS-CoV-2 Main Protease
手法: X-RAY DIFFRACTION / 解像度: 1.83 Å PDB-9bnz:
Crystal Structure of E166V SARS-CoV-2 Main Protease
手法: X-RAY DIFFRACTION / 解像度: 2.08 Å PDB-9bo0:
Crystal Structure of T21I SARS-CoV-2 Main Protease in Complex with Compound Mpro61
手法: X-RAY DIFFRACTION / 解像度: 2.16 Å PDB-9bo1:
Crystal Structure of T190I SARS-CoV-2 Main Protease in Complex with Compound Mpro61
手法: X-RAY DIFFRACTION / 解像度: 1.96 Å PDB-9bo2:
Crystal Structure of A173V/L50F SARS-CoV-2 Main Protease in Complex with Compound Mpro61
手法: X-RAY DIFFRACTION / 解像度: 1.91 Å PDB-9bo3:
Crystal Structure of E166V SARS-CoV-2 Main Protease in Complex with Compound Mpro61
手法: X-RAY DIFFRACTION / 解像度: 2.78 Å PDB-9bo4:
Crystal Structure of T21I SARS-CoV-2 Main Protease in Complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 2.28 Å PDB-9bo5:
Crystal Structure of T190I SARS-CoV-2 Main Protease in Complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 1.84 Å PDB-9bo6:
Crystal Structure of A173V/L50F SARS-CoV-2 Main Protease in Complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 1.61 Å PDB-9bo7:
Crystal Structure of L50F SARS-CoV-2 Main Protease in Complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 1.54 Å PDB-9bo8:
Crystal Structure of T21I SARS-CoV-2 Main Protease in Complex with GC376
手法: X-RAY DIFFRACTION / 解像度: 1.49 Å PDB-9bo9:
Crystal Structure of T190I SARS-CoV-2 Main Protease in Complex with GC376
手法: X-RAY DIFFRACTION / 解像度: 1.98 Å PDB-9boa:
Crystal Structure of A173V SARS-CoV-2 Main Protease in Complex with GC376
手法: X-RAY DIFFRACTION / 解像度: 1.7 Å PDB-9bob:
Crystal Structure of A173V/L50F SARS-CoV-2 Main Protease in Complex with GC376
手法: X-RAY DIFFRACTION / 解像度: 1.87 Å PDB-9boc:
Crystal Structure of L50F SARS-CoV-2 Main Protease in Complex with GC376
手法: X-RAY DIFFRACTION / 解像度: 1.68 Å PDB-9bod:
Crystal Structure of E166V/L50F SARS-CoV-2 Main Protease in Complex with GC376
手法: X-RAY DIFFRACTION / 解像度: 1.91 Å PDB-9boe:
Crystal Structure of E166V SARS-CoV-2 Main Protease in Complex with GC376
手法: X-RAY DIFFRACTION / 解像度: 2.02 Å PDB-9cdk:
SARS-CoV-2 Mpro A173V mutant in complex with small molecule inhibitor Mpro61
手法: X-RAY DIFFRACTION / 解像度: 1.61 Å PDB-9cdl:
SARS-CoV-2 Mpro E166V/L50F double mutant in complex with small molecule inhibitor Mpro61
手法: X-RAY DIFFRACTION / 解像度: 1.52 Å PDB-9cdm:
SARS-CoV-2 Mpro L50F mutant in complex with small molecule inhibitor Mpro61
手法: X-RAY DIFFRACTION / 解像度: 2.05 Å |
|---|
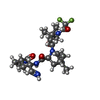
| 化合物 | ChemComp-4WI:
(1R,2S,5S)-N-{(1E,2S)-1-imino-3-[(3S)-2-oxopyrrolidin-3-yl]propan-2-yl}-6,6-dimethyl-3-[3-methyl-N-(trifluoroacetyl)-L-valyl]-3-azabicyclo[3.1.0]hexane-2-carboxamide / 薬剤, 抗ウイルス剤, プロテアーゼ阻害剤*YM
ChemComp-UED:
N~2~-[(benzyloxy)carbonyl]-N-{(2S)-1-hydroxy-3-[(3S)-2-oxopyrrolidin-3-yl]propan-2-yl}-L-leucinamide
|
|---|
| 由来 |   severe acute respiratory syndrome coronavirus 2 (ウイルス) severe acute respiratory syndrome coronavirus 2 (ウイルス)
|
|---|
 キーワード キーワード | VIRAL PROTEIN / Hydrolase / SARS-CoV-2 / Mpro / 3CL / main protease / inhibitor / small molecule / Mpro61 / drug resistant mutant / E166V / L50F / antiviral / mutant / small molecule inhibitor |
|---|
 著者
著者 リンク
リンク Acs Bio Med Chem Au /
Acs Bio Med Chem Au /  PubMed:39990941
PubMed:39990941




























 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




