-検索条件
-検索結果
検索 (著者・登録者: huang & kc)の結果全48件を表示しています

EMDB-29409: 
Full-length mouse 5-HT3A receptor in complex with ALB148471, pre-activated

EMDB-29410: 
Full-length mouse 5-HT3A receptor in complex with SMP100, pre-activated

EMDB-29411: 
Full-length mouse 5-HT3A receptor in complex with serotonin, pre-activated

EMDB-29418: 
Full-length mouse 5-HT3A receptor in complex with serotonin, open-like

EMDB-29421: 
Full-length mouse 5-HT3A receptor in complex with SMP100, open-like

EMDB-29422: 
Full-length mouse 5-HT3A receptor in complex with ALB148471, open-like

EMDB-35384: 
Cryo-EM structure of ATP13A2 in the E1-ATP state
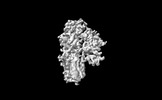
EMDB-35385: 
Cryo-EM structure of ATP13A2 in the E1-like state

EMDB-35386: 
Cryo-EM structure of ATP13A2 in the E2P state

EMDB-35387: 
Cryo-EM structure of ATP13A2 in the E2-Pi state

EMDB-35388: 
Cryo-EM structure of ATP13A2 in the nominal E1P state
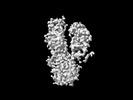
EMDB-35391: 
Cryo-EM structure of ATP13A2 in the putative of E2 state
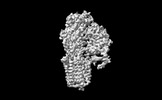
EMDB-35392: 
Cryo-EM structure of ATP13A2 in the E1P-ADP state

EMDB-35609: 
Cryo-EM structure of phosphoketolase from Bifidobacterium longum in octameric assembly

EMDB-35610: 
Cryo-EM structure of phosphoketolase from Bifidobacterium longum in dimeric assembly

EMDB-35611: 
Cryo-EM structure of cyanobacteria phosphoketolase complexed with AMPPNP in dimeric assembly

EMDB-35612: 
Cryo-EM structure of cyanobacteria phosphoketolase complexed with AMPPNPin dodecameric assembly

EMDB-35613: 
Cryo-EM structure of cyanobacteria phosphoketolase

EMDB-35617: 
Cryo-EM structure of cyanobacteria phosphoketolase in dodecameric assembly

EMDB-25419: 
Previously uncharacterized rectangular bacteria in the dolphin mouth

EMDB-28092: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-093

EMDB-28090: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-040

EMDB-28091: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-045
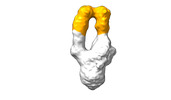
EMDB-28093: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-156
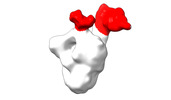
EMDB-28094: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-234

EMDB-28095: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-260

EMDB-28096: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-279

EMDB-28097: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-290
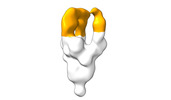
EMDB-28098: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-294

EMDB-28099: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-295

EMDB-28100: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-299

EMDB-28102: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-334
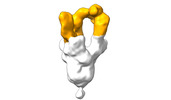
EMDB-28103: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-360

EMDB-28104: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-361

EMDB-28105: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-362

EMDB-28106: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-368

EMDB-28168: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-292

EMDB-28169: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-333
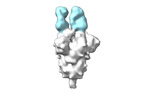
EMDB-28170: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-355

EMDB-28171: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-371

EMDB-25471: 
Structure of EBOV GP lacking the mucin-like domain with 1C11 scFv and 1C3 Fab bound

EMDB-11061: 
Structure of EV71 in complex with a protective antibody 38-3-11A Fab

EMDB-11062: 
Structure of EV71 in complex with a protective antibody 38-1-10A Fab

EMDB-21984: 
Structure of MZT1/GCP3-NHD and MZT1/GCP6-NHD in the gamma-TuRC lumenal bridge

EMDB-21985: 
Structure of MZT2/GCP-NHD and CDK5Rap2 at position 13 of the gamma-TuRC

EMDB-22221: 
SARS-CoV-2 HexaPro S One RBD up

EMDB-22222: 
SARS-CoV-2 HexaPro S Two RBD up

EMDB-20836: 
neurotensin receptor and arrestin2 complex
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
