+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8jd1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
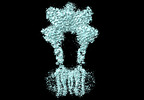
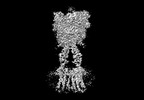
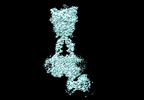
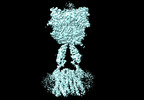
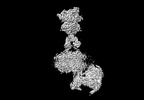
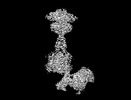
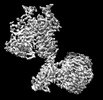
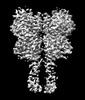
| タイトル | Cryo-EM structure of mGlu2-mGlu3 heterodimer in Rco state | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / Complex structure / mGlu2-3 heterodimer MEMBRANE PROTEIN (膜タンパク質) / Complex structure / mGlu2-3 heterodimer | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of cellular component organization / positive regulation of response to stimulus / regulation of response to drug / group II metabotropic glutamate receptor activity / response to chemical / regulation of cellular response to stress /  macrolide binding / macrolide binding /  TORC1 complex / TORC1 complex /  activin receptor binding / cytoplasmic side of membrane ...regulation of cellular component organization / positive regulation of response to stimulus / regulation of response to drug / group II metabotropic glutamate receptor activity / response to chemical / regulation of cellular response to stress / activin receptor binding / cytoplasmic side of membrane ...regulation of cellular component organization / positive regulation of response to stimulus / regulation of response to drug / group II metabotropic glutamate receptor activity / response to chemical / regulation of cellular response to stress /  macrolide binding / macrolide binding /  TORC1 complex / TORC1 complex /  activin receptor binding / cytoplasmic side of membrane / behavioral response to nicotine / intracellular glutamate homeostasis / signaling receptor inhibitor activity / activin receptor binding / cytoplasmic side of membrane / behavioral response to nicotine / intracellular glutamate homeostasis / signaling receptor inhibitor activity /  transforming growth factor beta receptor binding / TGFBR1 LBD Mutants in Cancer / type I transforming growth factor beta receptor binding / astrocyte projection / negative regulation of adenylate cyclase activity / G protein-coupled glutamate receptor signaling pathway / Class C/3 (Metabotropic glutamate/pheromone receptors) / negative regulation of activin receptor signaling pathway / heart trabecula formation / glutamate secretion / transforming growth factor beta receptor binding / TGFBR1 LBD Mutants in Cancer / type I transforming growth factor beta receptor binding / astrocyte projection / negative regulation of adenylate cyclase activity / G protein-coupled glutamate receptor signaling pathway / Class C/3 (Metabotropic glutamate/pheromone receptors) / negative regulation of activin receptor signaling pathway / heart trabecula formation / glutamate secretion /  terminal cisterna / terminal cisterna /  ryanodine receptor complex / ryanodine receptor complex /  I-SMAD binding / I-SMAD binding /  glutamate receptor activity / regulation of glutamate secretion / regulation of amyloid precursor protein catabolic process / long-term synaptic depression / protein maturation by protein folding / 'de novo' protein folding / ventricular cardiac muscle tissue morphogenesis / negative regulation of phosphoprotein phosphatase activity / postsynaptic modulation of chemical synaptic transmission / glutamate receptor activity / regulation of glutamate secretion / regulation of amyloid precursor protein catabolic process / long-term synaptic depression / protein maturation by protein folding / 'de novo' protein folding / ventricular cardiac muscle tissue morphogenesis / negative regulation of phosphoprotein phosphatase activity / postsynaptic modulation of chemical synaptic transmission /  FK506 binding / regulation of dopamine secretion / mTORC1-mediated signalling / TGF-beta receptor signaling activates SMADs / Calcineurin activates NFAT / calcium channel regulator activity / FK506 binding / regulation of dopamine secretion / mTORC1-mediated signalling / TGF-beta receptor signaling activates SMADs / Calcineurin activates NFAT / calcium channel regulator activity /  regulation of immune response / protein peptidyl-prolyl isomerization / supramolecular fiber organization / heart morphogenesis / regulation of ryanodine-sensitive calcium-release channel activity / regulation of immune response / protein peptidyl-prolyl isomerization / supramolecular fiber organization / heart morphogenesis / regulation of ryanodine-sensitive calcium-release channel activity /  regulation of synaptic transmission, glutamatergic / sarcoplasmic reticulum membrane / regulation of synaptic transmission, glutamatergic / sarcoplasmic reticulum membrane /  T細胞 / positive regulation of protein metabolic process / presynaptic modulation of chemical synaptic transmission / negative regulation of autophagy / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) / T細胞 / positive regulation of protein metabolic process / presynaptic modulation of chemical synaptic transmission / negative regulation of autophagy / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) /  筋小胞体 / response to cocaine / 筋小胞体 / response to cocaine /  プロリルイソメラーゼ / プロリルイソメラーゼ /  peptidyl-prolyl cis-trans isomerase activity / G protein-coupled receptor activity / calcium ion transmembrane transport / negative regulation of transforming growth factor beta receptor signaling pathway / Z disc / SARS-CoV-1 activates/modulates innate immune responses / peptidyl-prolyl cis-trans isomerase activity / G protein-coupled receptor activity / calcium ion transmembrane transport / negative regulation of transforming growth factor beta receptor signaling pathway / Z disc / SARS-CoV-1 activates/modulates innate immune responses /  regulation of protein localization / regulation of protein localization /  フォールディング / positive regulation of protein binding / フォールディング / positive regulation of protein binding /  presynaptic membrane / presynaptic membrane /  遺伝子発現 / G alpha (i) signalling events / protein refolding / 遺伝子発現 / G alpha (i) signalling events / protein refolding /  scaffold protein binding / chemical synaptic transmission / scaffold protein binding / chemical synaptic transmission /  postsynaptic membrane / positive regulation of canonical NF-kappaB signal transduction / transmembrane transporter binding / Potential therapeutics for SARS / amyloid fibril formation / postsynaptic membrane / positive regulation of canonical NF-kappaB signal transduction / transmembrane transporter binding / Potential therapeutics for SARS / amyloid fibril formation /  樹状突起スパイン / 樹状突起スパイン /  postsynaptic density / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / postsynaptic density / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  リン酸化 / リン酸化 /  神経繊維 / protein serine/threonine kinase activity / glutamatergic synapse / 神経繊維 / protein serine/threonine kinase activity / glutamatergic synapse /  樹状突起 / protein-containing complex binding / 樹状突起 / protein-containing complex binding /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Wang, X. / Wang, M. / Xu, T. / Feng, Y. / Han, S. / Lin, S. / Zhao, Q. / Wu, B. | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2023 ジャーナル: Cell Res / 年: 2023タイトル: Structural insights into dimerization and activation of the mGlu2-mGlu3 and mGlu2-mGlu4 heterodimers. 著者: Xinwei Wang / Mu Wang / Tuo Xu / Ye Feng / Qiang Shao / Shuo Han / Xiaojing Chu / Yechun Xu / Shuling Lin / Qiang Zhao / Beili Wu /  要旨: Heterodimerization of the metabotropic glutamate receptors (mGlus) has shown importance in the functional modulation of the receptors and offers potential drug targets for treating central nervous ...Heterodimerization of the metabotropic glutamate receptors (mGlus) has shown importance in the functional modulation of the receptors and offers potential drug targets for treating central nervous system diseases. However, due to a lack of molecular details of the mGlu heterodimers, understanding of the mechanisms underlying mGlu heterodimerization and activation is limited. Here we report twelve cryo-electron microscopy (cryo-EM) structures of the mGlu2-mGlu3 and mGlu2-mGlu4 heterodimers in different conformational states, including inactive, intermediate inactive, intermediate active and fully active conformations. These structures provide a full picture of conformational rearrangement of mGlu2-mGlu3 upon activation. The Venus flytrap domains undergo a sequential conformational change, while the transmembrane domains exhibit a substantial rearrangement from an inactive, symmetric dimer with diverse dimerization patterns to an active, asymmetric dimer in a conserved dimerization mode. Combined with functional data, these structures reveal that stability of the inactive conformations of the subunits and the subunit-G protein interaction pattern are determinants of asymmetric signal transduction of the heterodimers. Furthermore, a novel binding site for two mGlu4 positive allosteric modulators was observed in the asymmetric dimer interfaces of the mGlu2-mGlu4 heterodimer and mGlu4 homodimer, and may serve as a drug recognition site. These findings greatly extend our knowledge about signal transduction of the mGlus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8jd1.cif.gz 8jd1.cif.gz | 278.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8jd1.ent.gz pdb8jd1.ent.gz | 204.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8jd1.json.gz 8jd1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jd/8jd1 https://data.pdbj.org/pub/pdb/validation_reports/jd/8jd1 ftp://data.pdbj.org/pub/pdb/validation_reports/jd/8jd1 ftp://data.pdbj.org/pub/pdb/validation_reports/jd/8jd1 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  36172MC  8jcuC  8jcvC  8jcwC  8jcxC  8jcyC  8jczC  8jd0C  8jd2C  8jd3C  8jd4C  8jd5C  8jd6C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 109398.914 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: GRM2, GPRC1B, MGLUR2, FKBP1A, FKBP1, FKBP12 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GRM2, GPRC1B, MGLUR2, FKBP1A, FKBP1, FKBP12 / 発現宿主:  参照: UniProt: Q14416, UniProt: P62942,  プロリルイソメラーゼ プロリルイソメラーゼ | ||||
|---|---|---|---|---|---|
| #2: タンパク質 | 分子量: 112712.961 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: GRM3, GPRC1C, MGLUR3, MTOR / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GRM3, GPRC1C, MGLUR3, MTOR / 発現宿主:  参照: UniProt: Q14832, UniProt: A0A8V8TRG9,  non-specific serine/threonine protein kinase non-specific serine/threonine protein kinase | ||||
| #3: 化合物 |  グルタミン酸 グルタミン酸#4: 化合物 | ChemComp-CLR /  コレステロール コレステロール研究の焦点であるリガンドがあるか | Y | |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: mGlu2-mGlu3 heterodimer in presence of glutamate, JNJ-40411813, and CaCl2 タイプ: COMPLEX / Entity ID: #1-#2 / 由来: MULTIPLE SOURCES |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 800 nm Bright-field microscopy / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 800 nm |
| 撮影 | 電子線照射量: 70 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
3次元再構成 | 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 252188 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 精密化 | 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 51.45 Å2 | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj




















