+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8gj0 | ||||||
|---|---|---|---|---|---|---|---|
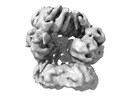
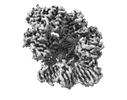
| タイトル | E. coli clamp loader with open clamp on primed template DNA (form 1) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA / clamp loader /  DNA clamp (DNAクランプ) / DNA clamp (DNAクランプ) /  AAA+ / AAA+ /  ATPase (ATPアーゼ) / DNA BINDING PROTEIN-DNA complex ATPase (ATPアーゼ) / DNA BINDING PROTEIN-DNA complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 DNA polymerase III, clamp loader complex / Hda-beta clamp complex / bacterial-type DNA replication / replication inhibiting complex / DNA clamp loader activity / DNA polymerase III, clamp loader complex / Hda-beta clamp complex / bacterial-type DNA replication / replication inhibiting complex / DNA clamp loader activity /  DNA polymerase III complex / DNA polymerase III complex /  replisome / regulation of DNA-templated DNA replication initiation / DNA strand elongation involved in DNA replication / DNA polymerase processivity factor activity ... replisome / regulation of DNA-templated DNA replication initiation / DNA strand elongation involved in DNA replication / DNA polymerase processivity factor activity ... DNA polymerase III, clamp loader complex / Hda-beta clamp complex / bacterial-type DNA replication / replication inhibiting complex / DNA clamp loader activity / DNA polymerase III, clamp loader complex / Hda-beta clamp complex / bacterial-type DNA replication / replication inhibiting complex / DNA clamp loader activity /  DNA polymerase III complex / DNA polymerase III complex /  replisome / regulation of DNA-templated DNA replication initiation / DNA strand elongation involved in DNA replication / DNA polymerase processivity factor activity / error-prone translesion synthesis / negative regulation of DNA-templated DNA replication initiation / 3'-5' exonuclease activity / ribonucleoside triphosphate phosphatase activity / response to radiation / DNA-templated DNA replication / replisome / regulation of DNA-templated DNA replication initiation / DNA strand elongation involved in DNA replication / DNA polymerase processivity factor activity / error-prone translesion synthesis / negative regulation of DNA-templated DNA replication initiation / 3'-5' exonuclease activity / ribonucleoside triphosphate phosphatase activity / response to radiation / DNA-templated DNA replication /  DNA複製 / DNA複製 /  DNAポリメラーゼ / DNAポリメラーゼ /  DNA-directed DNA polymerase activity / DNA-directed DNA polymerase activity /  DNA修復 / DNA damage response / protein homodimerization activity / DNA修復 / DNA damage response / protein homodimerization activity /  ATP hydrolysis activity / ATP hydrolysis activity /  DNA binding / DNA binding /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌)  Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Oakley, A.J. / Xu, Z.-Q. / Dixon, N.E. | ||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural characterisation of the complete cycle of sliding clamp loading in E. coli 著者: Xu, Z.-Q. / Oakley, A.J. / Dixon, N.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8gj0.cif.gz 8gj0.cif.gz | 1.2 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8gj0.ent.gz pdb8gj0.ent.gz | 816.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8gj0.json.gz 8gj0.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gj/8gj0 https://data.pdbj.org/pub/pdb/validation_reports/gj/8gj0 ftp://data.pdbj.org/pub/pdb/validation_reports/gj/8gj0 ftp://data.pdbj.org/pub/pdb/validation_reports/gj/8gj0 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  40081MC  8giyC  8gizC  8gj1C  8gj2C  8gj3C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-DNA polymerase III subunit ... , 4種, 6分子 ABCDEF
| #1: タンパク質 |  DNA polymerase III holoenzyme DNA polymerase III holoenzyme分子量: 38745.574 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: holA, b0640, JW0635 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: holA, b0640, JW0635 / 発現宿主:   Escherichia coli BL21 (大腸菌) / 参照: UniProt: P28630, Escherichia coli BL21 (大腸菌) / 参照: UniProt: P28630,  DNAポリメラーゼ DNAポリメラーゼ | ||||
|---|---|---|---|---|---|
| #2: タンパク質 |  DNA polymerase III holoenzyme / DNA polymerase III subunit gamma DNA polymerase III holoenzyme / DNA polymerase III subunit gamma分子量: 71228.648 Da / 分子数: 3 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: dnaX, dnaZ, dnaZX, b0470, JW0459 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: dnaX, dnaZ, dnaZX, b0470, JW0459 / 発現宿主:   Escherichia coli BL21 (大腸菌) / 参照: UniProt: P06710, Escherichia coli BL21 (大腸菌) / 参照: UniProt: P06710,  DNAポリメラーゼ DNAポリメラーゼ#3: タンパク質 | |  DNA polymerase III holoenzyme DNA polymerase III holoenzyme分子量: 36980.484 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: holB, b1099, JW1085 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: holB, b1099, JW1085 / 発現宿主:   Escherichia coli BL21 (大腸菌) / 参照: UniProt: P28631, Escherichia coli BL21 (大腸菌) / 参照: UniProt: P28631,  DNAポリメラーゼ DNAポリメラーゼ#4: タンパク質 | |  DNA polymerase III holoenzyme DNA polymerase III holoenzyme分子量: 15188.276 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: holD, b4372, JW4334 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: holD, b4372, JW4334 / 発現宿主:   Escherichia coli BL21 (大腸菌) / 参照: UniProt: P28632, Escherichia coli BL21 (大腸菌) / 参照: UniProt: P28632,  DNAポリメラーゼ DNAポリメラーゼ |
-タンパク質 , 1種, 2分子 HI
| #5: タンパク質 | 分子量: 40630.508 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: dnaN, b3701, JW3678 / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: dnaN, b3701, JW3678 / 発現宿主:   Escherichia coli BL21 (大腸菌) / 参照: UniProt: P0A988 Escherichia coli BL21 (大腸菌) / 参照: UniProt: P0A988 |
|---|
-DNA鎖 , 2種, 2分子 XY
| #6: DNA鎖 | 分子量: 7097.624 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|---|
| #7: DNA鎖 | 分子量: 20710.217 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-非ポリマー , 4種, 13分子 






| #8: 化合物 |  アデノシン二リン酸 アデノシン二リン酸#9: 化合物 | #10: 化合物 | #11: 化合物 | ChemComp-ZN / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: E. coli clamp loader with open clamp on primed template DNA タイプ: COMPLEX 詳細: Clamp loader complex composed of DNA polymerase III delta tau(3) delta' chi and psi subunits. Clamp composed of DNA polymerase III beta(2) subunits. Entity ID: #1-#7 / 由来: MULTIPLE SOURCES | |||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.419 MDa / 実験値: NO | |||||||||||||||||||||||||||||||||||||||||||||
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K-12 Escherichia coli (大腸菌) / 株: K-12 | |||||||||||||||||||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Escherichia coli (大腸菌) / 株: BL21 Escherichia coli (大腸菌) / 株: BL21 | |||||||||||||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.6 詳細: 30 mM Tris-HCl pH 7.6, 5 mM MgCl2, 2 mM ADP, 0.5 mM AlCl3, 5 mM NaF, 5 mM dithiothreitol, 0.25 mM EDTA, 2% glycerol. | |||||||||||||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| |||||||||||||||||||||||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: 30 microL of 6.0 microM delta/tau3/delta' complex was mixed with chi/psi complex, beta, and p/t DNA (annealed DNA oligonucleotides) at a molar ratio of 1:1.3 and dialysed at 4 degrees C ...詳細: 30 microL of 6.0 microM delta/tau3/delta' complex was mixed with chi/psi complex, beta, and p/t DNA (annealed DNA oligonucleotides) at a molar ratio of 1:1.3 and dialysed at 4 degrees C against 250 mL of 30 mM Tris-HCl pH 7.6, 5 mM MgCl2, 2 mM ADP, 0.5 mM AlCl3, 5 mM NaF, 5 mM dithiothreitol, 0.25 mM EDTA, 2% glycerol. | |||||||||||||||||||||||||||||||||||||||||||||
| 試料支持 | 詳細: Used a Zepto Low-pressure plasma systems (PLASMA CLEANER) (Diener Electronic) at 10% power for 120 seconds. グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: UltrAuFoil R1.2/1.3 | |||||||||||||||||||||||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 279 K 詳細: 3 uL of sample was applied onto a Ultrafoil Au R1.2/1.3 grid. Blot for 4.5 s with no extra force before plunging into liquid ethane. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1300 nm / 最小 デフォーカス(公称値): 400 nm / Cs Bright-field microscopy / 最大 デフォーカス(公称値): 1300 nm / 最小 デフォーカス(公称値): 400 nm / Cs : 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 6 sec. / 電子線照射量: 50 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 4226 |
| 電子光学装置 | エネルギーフィルター名称 : GIF Bioquantum / 詳細: unfiltered mode : GIF Bioquantum / 詳細: unfiltered mode |
| 画像スキャン | 横: 4096 / 縦: 4096 / 動画フレーム数/画像: 60 / 利用したフレーム数/画像: 1-60 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
CTF補正 | 詳細: CTF refinement per-particle was performed in RELION before final 3D reconstruction. タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1433000 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 75000 / アルゴリズム: BACK PROJECTION / クラス平均像の数: 2 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 136.43 Å2 | ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj

















































