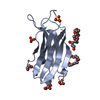
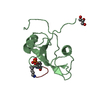
登録情報 データベース : PDB / ID : 6bubタイトル Crystal structure of the PI3KC2alpha PX domain in space group P432 Phosphatidylinositol 4-phosphate 3-kinase C2 domain-containing subunit alpha キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 2.604 Å データ登録者 Chen, K.-E. / Collins, B.M. ジャーナル : Structure / 年 : 2018タイトル : Molecular Basis for Membrane Recruitment by the PX and C2 Domains of Class II Phosphoinositide 3-Kinase-C2α.著者 : Kai-En Chen / Vikas A Tillu / Mintu Chandra / Brett M Collins / 要旨 : Phosphorylation of phosphoinositides by the class II phosphatidylinositol 3-kinase (PI3K) PI3K-C2α is essential for many processes, including neuroexocytosis and formation of clathrin-coated ... Phosphorylation of phosphoinositides by the class II phosphatidylinositol 3-kinase (PI3K) PI3K-C2α is essential for many processes, including neuroexocytosis and formation of clathrin-coated vesicles. A defining feature of the class II PI3Ks is a C-terminal module composed of phox-homology (PX) and C2 membrane interacting domains; however, the mechanisms that control their specific cellular localization remain poorly understood. Here we report the crystal structure of the C2 domain of PI3K-C2α in complex with the phosphoinositide head-group mimic inositol hexaphosphate, revealing two distinct pockets for membrane binding. The C2 domain preferentially binds to phosphatidylinositol 4,5-bisphosphate and phosphatidylinositol (3,4,5)-trisphosphate, and low-resolution structures of the combined PX-C2 module by small-angle X-ray scattering reveal a compact conformation in which cooperative lipid binding by each domain binding can occur. Finally, we demonstrate an unexpected role for calcium in perturbing the membrane interactions of the PX-C2 module, which we speculate may be important for regulating the activity of PI3K-C2α. 履歴 登録 2017年12月9日 登録サイト / 処理サイト 改定 1.0 2018年10月17日 Provider / タイプ 改定 1.1 2018年12月19日 Group / Database references / カテゴリ / Item / _citation.page_first改定 1.2 2023年10月4日 Group Advisory / Data collection ... Advisory / Data collection / Database references / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_unobs_or_zero_occ_atoms Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSFERASE (転移酵素) /
TRANSFERASE (転移酵素) /  PX domain /
PX domain /  lipid binding (脂質) /
lipid binding (脂質) /  phosphoinositide (ホスファチジルイノシトール) / PI3-kinase
phosphoinositide (ホスファチジルイノシトール) / PI3-kinase 機能・相同性情報
機能・相同性情報 phosphatidylinositol-4-phosphate 3-kinase / clathrin coat assembly / membrane organization / phosphatidylinositol biosynthetic process /
phosphatidylinositol-4-phosphate 3-kinase / clathrin coat assembly / membrane organization / phosphatidylinositol biosynthetic process /  phosphatidylinositol 3-kinase complex / 1-phosphatidylinositol-4-phosphate 3-kinase activity ...vascular associated smooth muscle contraction / Synthesis of PIPs at the late endosome membrane / Synthesis of PIPs at the early endosome membrane / Synthesis of PIPs at the Golgi membrane /
phosphatidylinositol 3-kinase complex / 1-phosphatidylinositol-4-phosphate 3-kinase activity ...vascular associated smooth muscle contraction / Synthesis of PIPs at the late endosome membrane / Synthesis of PIPs at the early endosome membrane / Synthesis of PIPs at the Golgi membrane /  phosphatidylinositol-4-phosphate 3-kinase / clathrin coat assembly / membrane organization / phosphatidylinositol biosynthetic process /
phosphatidylinositol-4-phosphate 3-kinase / clathrin coat assembly / membrane organization / phosphatidylinositol biosynthetic process /  phosphatidylinositol 3-kinase complex / 1-phosphatidylinositol-4-phosphate 3-kinase activity / 1-phosphatidylinositol-4,5-bisphosphate 3-kinase activity /
phosphatidylinositol 3-kinase complex / 1-phosphatidylinositol-4-phosphate 3-kinase activity / 1-phosphatidylinositol-4,5-bisphosphate 3-kinase activity /  phosphatidylinositol-4,5-bisphosphate 3-kinase /
phosphatidylinositol-4,5-bisphosphate 3-kinase /  PI3キナーゼ /
PI3キナーゼ /  クラスリン / phosphatidylinositol-3-phosphate biosynthetic process / 1-phosphatidylinositol-3-kinase activity /
クラスリン / phosphatidylinositol-3-phosphate biosynthetic process / 1-phosphatidylinositol-3-kinase activity /  clathrin binding / positive regulation of cell migration involved in sprouting angiogenesis / Golgi Associated Vesicle Biogenesis /
clathrin binding / positive regulation of cell migration involved in sprouting angiogenesis / Golgi Associated Vesicle Biogenesis /  エキソサイトーシス / Synthesis of PIPs at the plasma membrane / platelet-derived growth factor receptor signaling pathway / positive regulation of autophagy /
エキソサイトーシス / Synthesis of PIPs at the plasma membrane / platelet-derived growth factor receptor signaling pathway / positive regulation of autophagy /  phosphatidylinositol binding / phosphatidylinositol 3-kinase/protein kinase B signal transduction / epidermal growth factor receptor signaling pathway /
phosphatidylinositol binding / phosphatidylinositol 3-kinase/protein kinase B signal transduction / epidermal growth factor receptor signaling pathway /  ゴルジ体 /
ゴルジ体 /  エンドサイトーシス / insulin receptor signaling pathway /
エンドサイトーシス / insulin receptor signaling pathway /  Clathrin-mediated endocytosis / vesicle /
Clathrin-mediated endocytosis / vesicle /  リン酸化 / intracellular membrane-bounded organelle / extracellular exosome /
リン酸化 / intracellular membrane-bounded organelle / extracellular exosome /  核質 /
核質 /  ATP binding /
ATP binding /  生体膜 /
生体膜 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.604 Å
分子置換 / 解像度: 2.604 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Structure / 年: 2018
ジャーナル: Structure / 年: 2018
 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6bub.cif.gz
6bub.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6bub.ent.gz
pdb6bub.ent.gz PDB形式
PDB形式 6bub.json.gz
6bub.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/bu/6bub
https://data.pdbj.org/pub/pdb/validation_reports/bu/6bub ftp://data.pdbj.org/pub/pdb/validation_reports/bu/6bub
ftp://data.pdbj.org/pub/pdb/validation_reports/bu/6bub リンク
リンク 集合体
集合体
 要素
要素
 Homo sapiens (ヒト) / 遺伝子: PIK3C2A / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PIK3C2A / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) phosphatidylinositol-4-phosphate 3-kinase
phosphatidylinositol-4-phosphate 3-kinase 硫酸塩
硫酸塩 グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Australian Synchrotron
Australian Synchrotron  / ビームライン: MX2 / 波長: 1 Å
/ ビームライン: MX2 / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj








