+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3wao | ||||||
|---|---|---|---|---|---|---|---|
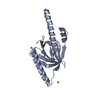
| タイトル | Crystal structure of Atg13 LIR-fused human LC3B_2-119 | ||||||
 要素 要素 | Autophagy-related protein 13, Microtubule-associated proteins 1A/1B light chain 3B | ||||||
 キーワード キーワード |  APOPTOSIS (アポトーシス) / UBIQUITIN-LIKE FOLD / APOPTOSIS (アポトーシス) / UBIQUITIN-LIKE FOLD /  AUTOPHAGY (オートファジー) AUTOPHAGY (オートファジー) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 regulation of protein lipidation / SARS-CoV-2 modulates autophagy / Atg1/ULK1 kinase complex / response to mitochondrial depolarisation / regulation of protein lipidation / SARS-CoV-2 modulates autophagy / Atg1/ULK1 kinase complex / response to mitochondrial depolarisation /  ceramide binding / protein localization to phagophore assembly site / phagophore assembly site membrane / piecemeal microautophagy of the nucleus / cellular response to nitrogen starvation / ceramide binding / protein localization to phagophore assembly site / phagophore assembly site membrane / piecemeal microautophagy of the nucleus / cellular response to nitrogen starvation /  phosphatidylethanolamine binding ... phosphatidylethanolamine binding ... regulation of protein lipidation / SARS-CoV-2 modulates autophagy / Atg1/ULK1 kinase complex / response to mitochondrial depolarisation / regulation of protein lipidation / SARS-CoV-2 modulates autophagy / Atg1/ULK1 kinase complex / response to mitochondrial depolarisation /  ceramide binding / protein localization to phagophore assembly site / phagophore assembly site membrane / piecemeal microautophagy of the nucleus / cellular response to nitrogen starvation / ceramide binding / protein localization to phagophore assembly site / phagophore assembly site membrane / piecemeal microautophagy of the nucleus / cellular response to nitrogen starvation /  phosphatidylethanolamine binding / protein kinase regulator activity / phagophore assembly site / Translation of Replicase and Assembly of the Replication Transcription Complex / TBC/RABGAPs / positive regulation of protein targeting to mitochondrion / phosphatidylethanolamine binding / protein kinase regulator activity / phagophore assembly site / Translation of Replicase and Assembly of the Replication Transcription Complex / TBC/RABGAPs / positive regulation of protein targeting to mitochondrion /  オートファジー / Receptor Mediated Mitophagy / オートファジー / Receptor Mediated Mitophagy /  axoneme / autophagosome membrane / organelle membrane / autophagosome maturation / axoneme / autophagosome membrane / organelle membrane / autophagosome maturation /  マイトファジー / マイトファジー /  autophagosome assembly / autophagosome assembly /  オートファゴソーム / オートファゴソーム /  細胞内膜系 / positive regulation of autophagy / PINK1-PRKN Mediated Mitophagy / Pexophagy / cellular response to starvation / protein serine/threonine kinase activator activity / 細胞内膜系 / positive regulation of autophagy / PINK1-PRKN Mediated Mitophagy / Pexophagy / cellular response to starvation / protein serine/threonine kinase activator activity /  ミトコンドリア / ミトコンドリア /  オートファジー / オートファジー /  オートファジー / KEAP1-NFE2L2 pathway / Translation of Replicase and Assembly of the Replication Transcription Complex / cytoplasmic vesicle / オートファジー / KEAP1-NFE2L2 pathway / Translation of Replicase and Assembly of the Replication Transcription Complex / cytoplasmic vesicle /  microtubule binding / microtubule binding /  微小管 / negative regulation of cell population proliferation / intracellular membrane-bounded organelle / 微小管 / negative regulation of cell population proliferation / intracellular membrane-bounded organelle /  ubiquitin protein ligase binding / endoplasmic reticulum membrane / ubiquitin protein ligase binding / endoplasmic reticulum membrane /  protein kinase binding / protein kinase binding /  ミトコンドリア / ミトコンドリア /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.6 Å 分子置換 / 解像度: 2.6 Å | ||||||
 データ登録者 データ登録者 | Suzuki, H. / Tabata, K. / Morita, E. / Kawasaki, M. / Kato, R. / Dobson, R.C.J. / Yoshimori, T. / Wakatsuki, S. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2014 ジャーナル: Structure / 年: 2014タイトル: Structural basis of the autophagy-related LC3/Atg13 LIR complex: recognition and interaction mechanism. 著者: Suzuki, H. / Tabata, K. / Morita, E. / Kawasaki, M. / Kato, R. / Dobson, R.C. / Yoshimori, T. / Wakatsuki, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3wao.cif.gz 3wao.cif.gz | 211 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3wao.ent.gz pdb3wao.ent.gz | 172.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3wao.json.gz 3wao.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/wa/3wao https://data.pdbj.org/pub/pdb/validation_reports/wa/3wao ftp://data.pdbj.org/pub/pdb/validation_reports/wa/3wao ftp://data.pdbj.org/pub/pdb/validation_reports/wa/3wao | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 15555.693 Da / 分子数: 4 / 断片: RESIDUES 436-447, RESIDUES 2-119 / 由来タイプ: 組換発現 詳細: THE FUSION PROTEIN OF AUTOPHAGY-RELATED GENE 13 LIR (RESIDUES 436-447), LINKER (GLY SER) AND MICROTUBULE-ASSOCIATED PROTEINS 1A/1B LIGHT CHAIN 3B (RESIDUES 2-119) 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: MAP1LC3B, MAP1ALC3 / プラスミド: pET30 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MAP1LC3B, MAP1ALC3 / プラスミド: pET30 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O75143, UniProt: Q9GZQ8 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: O75143, UniProt: Q9GZQ8 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.18 Å3/Da / 溶媒含有率: 43.66 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 0.1M Sodium citrate tribasic dihydrate 10% PEG 3350, VAPOR DIFFUSION, HANGING DROP, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 95 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Photon Factory Photon Factory  / ビームライン: BL-17A / 波長: 0.98 Å / ビームライン: BL-17A / 波長: 0.98 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2012年1月22日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.98 Å / 相対比: 1 : 0.98 Å / 相対比: 1 |
| 反射 | 解像度: 2.6→43.44 Å / Num. obs: 16435 / % possible obs: 99.9 % / Rmerge(I) obs: 0.068 / Net I/σ(I): 14.4 |
| 反射 シェル | 解像度: 2.6→2.74 Å / Rmerge(I) obs: 0.74 / Mean I/σ(I) obs: 2.4 / Num. unique all: 2372 / % possible all: 99.5 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 3VTU 解像度: 2.6→43.44 Å / Cor.coef. Fo:Fc: 0.94 / Cor.coef. Fo:Fc free: 0.883 / SU B: 37.124 / SU ML: 0.349 / 交差検証法: THROUGHOUT / ESU R Free: 0.4 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 61.477 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.6→43.44 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.6→2.667 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj








