登録情報 データベース : PDB / ID : 3qduタイトル Structure of Boletus edulis lectin in complex with N,N-diacetyl chitobiose BOLETUS EDULIS LECTIN キーワード / / / / / / / / 機能・相同性 / / / / / / / / 生物種 Boletus edulis (ヤマドリタケ)手法 / / / 解像度 : 2 Å データ登録者 Bovi, M. / Carrizo, M.E. / Capaldi, S. / Perduca, M. / Chiarelli, L.R. / Galliano, M. / Monaco, H.L. ジャーナル : Glycobiology / 年 : 2011タイトル : Structure of a lectin with antitumoral properties in king bolete (Boletus edulis) mushrooms.著者 : Bovi, M. / Carrizo, M.E. / Capaldi, S. / Perduca, M. / Chiarelli, L.R. / Galliano, M. / Monaco, H.L. 履歴 登録 2011年1月19日 登録サイト / 処理サイト 改定 1.0 2011年2月23日 Provider / タイプ 改定 1.1 2011年7月13日 Group 改定 1.2 2011年11月2日 Group 改定 2.0 2020年7月29日 Group Advisory / Atomic model ... Advisory / Atomic model / Data collection / Derived calculations / Non-polymer description / Refinement description / Structure summary カテゴリ atom_site / atom_site_anisotrop ... atom_site / atom_site_anisotrop / chem_comp / database_PDB_caveat / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_unobs_or_zero_occ_atoms / pdbx_validate_chiral / refine / refine_ls_restr / refine_ls_shell / reflns / software / struct_asym / struct_conn / struct_site / struct_site_gen Item _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ... _atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site_anisotrop.U[1][1] / _atom_site_anisotrop.U[1][2] / _atom_site_anisotrop.U[1][3] / _atom_site_anisotrop.U[2][2] / _atom_site_anisotrop.U[2][3] / _atom_site_anisotrop.U[3][3] / _atom_site_anisotrop.pdbx_auth_asym_id / _atom_site_anisotrop.pdbx_auth_atom_id / _atom_site_anisotrop.pdbx_auth_comp_id / _atom_site_anisotrop.pdbx_auth_seq_id / _atom_site_anisotrop.pdbx_label_asym_id / _atom_site_anisotrop.pdbx_label_atom_id / _atom_site_anisotrop.pdbx_label_comp_id / _chem_comp.formula / _chem_comp.formula_weight / _chem_comp.id / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.pdbx_number_of_molecules / _entity.type / _pdbx_struct_assembly_gen.asym_id_list / _refine.B_iso_mean / _refine.details / _refine.ls_R_factor_R_free / _refine.ls_R_factor_R_work / _refine.ls_R_factor_obs / _refine.ls_number_reflns_all / _refine.ls_percent_reflns_obs / _refine.pdbx_starting_model / _refine_ls_shell.d_res_low / _reflns.observed_criterion_sigma_F / _software.classification / _software.name / _software.version / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_value_order / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id 解説 / Provider / タイプ 改定 2.1 2023年9月13日 Group Data collection / Database references ... Data collection / Database references / Refinement description / Structure summary カテゴリ chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_DOI / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード Boletus edulis (ヤマドリタケ) /
Boletus edulis (ヤマドリタケ) /  lectin (レクチン) /
lectin (レクチン) /  mushroom (キノコ) / T-antigen disaccharide / N / N-diacetyl chitobiose /
mushroom (キノコ) / T-antigen disaccharide / N / N-diacetyl chitobiose /  Carbohydrate (炭水化物) /
Carbohydrate (炭水化物) /  Sugar Binding (砂糖)
Sugar Binding (砂糖) carbohydrate binding /
carbohydrate binding /  サンドイッチ / Mainly Beta / Boletus edulis lectin
サンドイッチ / Mainly Beta / Boletus edulis lectin 機能・相同性情報
機能・相同性情報
 Boletus edulis (ヤマドリタケ)
Boletus edulis (ヤマドリタケ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Glycobiology / 年: 2011
ジャーナル: Glycobiology / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3qdu.cif.gz
3qdu.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3qdu.ent.gz
pdb3qdu.ent.gz PDB形式
PDB形式 3qdu.json.gz
3qdu.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/qd/3qdu
https://data.pdbj.org/pub/pdb/validation_reports/qd/3qdu ftp://data.pdbj.org/pub/pdb/validation_reports/qd/3qdu
ftp://data.pdbj.org/pub/pdb/validation_reports/qd/3qdu リンク
リンク 集合体
集合体
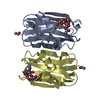

 要素
要素
 Boletus edulis (ヤマドリタケ) / 参照: UniProt: F2Z266*PLUS
Boletus edulis (ヤマドリタケ) / 参照: UniProt: F2Z266*PLUS オリゴ糖 / 分子量: 424.401 Da / 分子数: 4 / 由来タイプ: 組換発現
オリゴ糖 / 分子量: 424.401 Da / 分子数: 4 / 由来タイプ: 組換発現 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-1 / 波長: 0.934 Å
/ ビームライン: ID14-1 / 波長: 0.934 Å : 0.934 Å / 相対比: 1
: 0.934 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj