+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30869 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the multiple peptide resistance factor (MprF) loaded with two lysyl-phosphatidylglycerol molecules | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | bacteria membrane protein /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||||||||
| 機能・相同性 | Phosphatidylglycerol lysyltransferase, C-terminal / Phosphatidylglycerol lysyltransferase, C-terminal / Acyl-CoA N-acyltransferase /  細胞膜 / Bifunctional lysylphosphatidylglycerol flippase/synthetase MprF 細胞膜 / Bifunctional lysylphosphatidylglycerol flippase/synthetase MprF 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Rhizobium tropici (根粒菌) Rhizobium tropici (根粒菌) | ||||||||||||
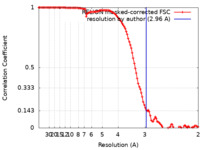
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.96 Å クライオ電子顕微鏡法 / 解像度: 2.96 Å | ||||||||||||
 データ登録者 データ登録者 | Song DF / Jiao HZ | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Phospholipid translocation captured in a bifunctional membrane protein MprF. 著者: Danfeng Song / Haizhan Jiao / Zhenfeng Liu /  要旨: As a large family of membrane proteins crucial for bacterial physiology and virulence, the Multiple Peptide Resistance Factors (MprFs) utilize two separate domains to synthesize and translocate ...As a large family of membrane proteins crucial for bacterial physiology and virulence, the Multiple Peptide Resistance Factors (MprFs) utilize two separate domains to synthesize and translocate aminoacyl phospholipids to the outer leaflets of bacterial membranes. The function of MprFs enables Staphylococcus aureus and other pathogenic bacteria to acquire resistance to daptomycin and cationic antimicrobial peptides. Here we present cryo-electron microscopy structures of MprF homodimer from Rhizobium tropici (RtMprF) at two different states in complex with lysyl-phosphatidylglycerol (LysPG). RtMprF contains a membrane-embedded lipid-flippase domain with two deep cavities opening toward the inner and outer leaflets of the membrane respectively. Intriguingly, a hook-shaped LysPG molecule is trapped inside the inner cavity with its head group bent toward the outer cavity which hosts a second phospholipid-binding site. Moreover, RtMprF exhibits multiple conformational states with the synthase domain adopting distinct positions relative to the flippase domain. Our results provide a detailed framework for understanding the mechanisms of MprF-mediated modification and translocation of phospholipids. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30869.map.gz emd_30869.map.gz | 8.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30869-v30.xml emd-30869-v30.xml emd-30869.xml emd-30869.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30869_fsc.xml emd_30869_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30869.png emd_30869.png | 126.8 KB | ||
| Filedesc metadata |  emd-30869.cif.gz emd-30869.cif.gz | 7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30869 http://ftp.pdbj.org/pub/emdb/structures/EMD-30869 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30869 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30869 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30869.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30869.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : bacterial membrane protein
| 全体 | 名称: bacterial membrane protein |
|---|---|
| 要素 |
|
-超分子 #1: bacterial membrane protein
| 超分子 | 名称: bacterial membrane protein / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: A recombinant protein from Rhizobium tropici expressed in Escherichia coli cells |
|---|---|
| 由来(天然) | 生物種:   Rhizobium tropici (根粒菌) Rhizobium tropici (根粒菌) |
-分子 #1: Bifunctional lysylphosphatidylglycerol flippase/synthetase MprF
| 分子 | 名称: Bifunctional lysylphosphatidylglycerol flippase/synthetase MprF タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rhizobium tropici (根粒菌) Rhizobium tropici (根粒菌) |
| 分子量 | 理論値: 96.094672 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSSPIDLESA DEELEERGGI VGLLNRYRAL IVAVLTVAVF CIAAYAIYDL TTEVRYDDVV HALTTTKISS VLLALLFTGL SFASLIFYD QNALEYIGKR LPFPHVALTS FSAYAVGNTA GFGALSAGAI RYRAYTRLGL SPDDITRVIA FVTLAFGLGL A SVGAMALL ...文字列: MSSPIDLESA DEELEERGGI VGLLNRYRAL IVAVLTVAVF CIAAYAIYDL TTEVRYDDVV HALTTTKISS VLLALLFTGL SFASLIFYD QNALEYIGKR LPFPHVALTS FSAYAVGNTA GFGALSAGAI RYRAYTRLGL SPDDITRVIA FVTLAFGLGL A SVGAMALL VIADEIGPLI SVDGLWLRLI AIAILAALAF VVYAGRNGRE VRIGPVAVRL PDSRTWSRQF LVTAFDIAAS AS VLYVLLP ETSIGWPGFF AIYAIAVGLG VLSHVPAGFG VFETIIIAWL GSSVNEDAVL SSLVLYRVIY NVIPLVIAIA AIS VAELRR FVDHPVASSM RRIGARLMPQ LLSAFALLLG MMLVFSSVTP TPDHNLIVLS DYLPLSLVES AHFLSSLLGL AIIV AARGL SQRLDGAWWV STFSALFALF FSLLKAIAIV EAGLLAFFVF SLVVSRRLFK RPASLLNQTL TAGWLTAIAV VCIGA IVVL FFVYRDVGYS NELWWQFEFA DEAPRGLRAA LGISIVSSAI AIFSLLRPAT KRPEPVSDDA VARAVEIVRK QGVADA NLV RMGDKSIMFS EKGDAFIMYG KQGRSWIALF DPVGPRQALP DLIWRFVETA RAAGCRSVFY QISPALLSYC ADAGLRA FK LGELAVVNLA NFELKGGKWA NLRQTASRAV RDGLEFAVIE PQDIPDVLDQ LAHVSDTWLA DHNAKEKSFS LGAFDPDY V CSQPVGVLKK DGKIVAFANI LMTETKEEGS VDLMRFSPDA PKGSMDFLFV QILEYLKGEG FQRFNLGMAP LSGMSRRES APVWDRVGGT VFEHGERFYN FKGLRAFKSK FHPEWQPRYL AVSGGVSPMI ALMDATFLIG GGLKGVVRKK LAAALEHHHH HH UniProtKB: Bifunctional lysylphosphatidylglycerol flippase/synthetase MprF |
-分子 #2: DODECYL-BETA-D-MALTOSIDE
| 分子 | 名称: DODECYL-BETA-D-MALTOSIDE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: LMT |
|---|---|
| 分子量 | 理論値: 510.615 Da |
| Chemical component information |  ChemComp-LMT: |
-分子 #3: [(2~{R})-3-[[(2~{S})-3-[(2~{S})-2,6-bis(azanyl)hexanoyl]oxy-2-oxi...
| 分子 | 名称: [(2~{R})-3-[[(2~{S})-3-[(2~{S})-2,6-bis(azanyl)hexanoyl]oxy-2-oxidanyl-propoxy]-oxidanyl-phosphoryl]oxy-2-hexadecanoyloxy-propyl] hexadecanoate タイプ: ligand / ID: 3 / コピー数: 4 / 式: EV9 |
|---|---|
| 分子量 | 理論値: 851.142 Da |
| Chemical component information |  ChemComp-EV9: |
-分子 #4: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
| 分子 | 名称: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE / タイプ: ligand / ID: 4 / コピー数: 2 / 式: LHG |
|---|---|
| 分子量 | 理論値: 722.97 Da |
| Chemical component information |  ChemComp-LHG: |
-分子 #5: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
| 分子 | 名称: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-1-[(PALMITOYLOXY)METHYL]ETHYL STEARATE タイプ: ligand / ID: 5 / コピー数: 2 / 式: PGT |
|---|---|
| 分子量 | 理論値: 751.023 Da |
| Chemical component information |  ChemComp-PGT: |
-分子 #6: (2~{R},3~{S},4~{S},5~{S},6~{S})-2-(hydroxymethyl)-6-[(2~{R},3~{S}...
| 分子 | 名称: (2~{R},3~{S},4~{S},5~{S},6~{S})-2-(hydroxymethyl)-6-[(2~{R},3~{S},4~{R},5~{R},6~{R})-2-(hydroxymethyl)-6-[2-[[(2~{R},3~{S},4~{R},5~{R},6~{S})-6-(hydroxymethyl)-5- ...名称: (2~{R},3~{S},4~{S},5~{S},6~{S})-2-(hydroxymethyl)-6-[(2~{R},3~{S},4~{R},5~{R},6~{R})-2-(hydroxymethyl)-6-[2-[[(2~{R},3~{S},4~{R},5~{R},6~{S})-6-(hydroxymethyl)-5-[(2~{S},3~{R},4~{S},5~{S},6~{R})-6-(hydroxymethyl)-3,4,5-tris(oxidanyl)oxan-2-yl]oxy-3,4-bis(oxidanyl)oxan-2-yl]oxymethyl]-4-[(1~{R},2~{R},4~{S},5'~{R},6~{R},7~{R},8~{R},9~{S},12~{S},13~{R},16~{S})-5',7,9,13-tetramethylspiro[5-oxapentacyclo[10.8.0.0^{2,9}.0^{4,8}.0^{13,18}]icos-18-ene-6,2'-oxane]-16-yl]oxy-butoxy]-4,5-bis(oxidanyl)oxan-3-yl]oxy-oxane-3,4,5-triol タイプ: ligand / ID: 6 / コピー数: 2 / 式: J4U |
|---|---|
| 分子量 | 理論値: 1.165315 KDa |
| Chemical component information |  ChemComp-J4U: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 2 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 13 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | The protein is reconstituted in lipid nanodiscs |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-32 / 撮影したグリッド数: 1 / 実像数: 2579 / 平均露光時間: 5.2 sec. / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7duw: |
 ムービー
ムービー コントローラー
コントローラー