-検索条件
-検索結果
検索 (著者・登録者: yu & lt)の結果348件中、1から50件目までを表示しています

EMDB-18658: 
Structure of the NCOA4 (Nuclear Receptor Coactivator 4)-FTH1 (H-Ferritin) complex

PDB-8qu9: 
Structure of the NCOA4 (Nuclear Receptor Coactivator 4)-FTH1 (H-Ferritin) complex

EMDB-18373: 
cryo-EM structure of apo Clostridioides difficile toxin B

EMDB-18374: 
cryo-EM structure complex of Frizzled-7 and Clostridioides difficile toxin B

PDB-8qen: 
cryo-EM structure of apo Clostridioides difficile toxin B

PDB-8qeo: 
cryo-EM structure complex of Frizzled-7 and Clostridioides difficile toxin B

EMDB-41816: 
Cryo-EM structure of the RAF1-HSP90-CDC37 complex in the closed state

EMDB-41817: 
Cryo-EM structure of the HSP90 dimer (NTD-MD) in the semi-open state

EMDB-41818: 
Cryo-EM structure of the cross-linked HSP90 dimer (NTD-MD) in the semi-open state

PDB-8u1l: 
Cryo-EM structure of the RAF1-HSP90-CDC37 complex in the closed state

PDB-8u1m: 
Cryo-EM structure of the HSP90 dimer (NTD-MD) in the semi-open state

PDB-8u1n: 
Cryo-EM structure of the cross-linked HSP90 dimer (NTD-MD) in the semi-open state

EMDB-16229: 
Cryo-EM structure of the bacterial replication origin opening basal unwinding system

PDB-8btg: 
Cryo-EM structure of the bacterial replication origin opening basal unwinding system

EMDB-37631: 
Hepatitis B virus capsid (HBV core protein)

EMDB-37634: 
SR protein kinase 2 bound at 2-fold vertex of Hepatitis B virus capsid

EMDB-38062: 
Hepatitis B virus capsid in complex with SR protein kinase 2

EMDB-41830: 
Lipidated recombinant apolipoprotein E4

EMDB-41831: 
Gradient-fixed lipidated recombinant apolipoprotein E4

EMDB-17208: 
CRYO-EM STRUCTURE OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : PARENTAL STRAIN

EMDB-17212: 
CRYO-EM STRUCTURE OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : TB11CS6H1 snoRNA mutant

EMDB-17249: 
CRYO-EM CONSENSUS MAP OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : PARENTAL STRAIN

EMDB-17250: 
CRYO-EM FOCUSED REFINEMENT MAPS OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : PARENTAL STRAIN

EMDB-17254: 
CRYO-EM CONSENSUS MAP OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : TB11CS6H1 SKO

EMDB-17255: 
CRYO-EM FOCUSED REFINEMENT OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : TB11CS6H1 snoRNA mutant

PDB-8ova: 
CRYO-EM STRUCTURE OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : PARENTAL STRAIN

PDB-8ove: 
CRYO-EM STRUCTURE OF TRYPANOSOMA BRUCEI PROCYCLIC FORM 80S RIBOSOME : TB11CS6H1 snoRNA mutant

EMDB-41075: 
SARS-CoV-2 spike in complex with Fab 71281-33

EMDB-41076: 
SARS-CoV-2 spike in complex with Fab 71281-33 (2)

EMDB-29281: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

EMDB-29282: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53

PDB-8flk: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

PDB-8flm: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53

EMDB-34715: 
Cryo-EM structure of ComA bound to its mature substrate CSP peptide
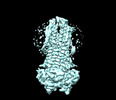
EMDB-36882: 
Cryo-EM structure of nucleotide-bound ComA with ZinC ion

EMDB-36936: 
Cryo-EM structure of nucleotide-bound ComA E647Q mutant with Mg2+

EMDB-34712: 
Cryo-EM structure of nucleotide-bound ComA at outward-facing state with EC gate closed conformation

EMDB-34713: 
Cryo-EM structure of nucleotide-bound ComA at outward-facing state with EC gate open conformation

EMDB-34714: 
Cryo-EM structure of nucleotide-bound ComA E647Q mutant

EMDB-34716: 
Cryo-EM structure of ComC bound ComA C17A at inward-facing state

EMDB-40184: 
Structure of the Spizellomyces punctatus Fanzor (SpuFz) in complex with omega RNA and target DNA

PDB-8gkh: 
Structure of the Spizellomyces punctatus Fanzor (SpuFz) in complex with omega RNA and target DNA
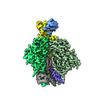
EMDB-29248: 
Cryo-EM Structure of PG9RSH DU011 Fab in complex with BG505 DS-SOSIP.664
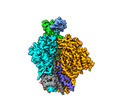
EMDB-29264: 
Cryo-EM Structure of PG9RSH DU025 Fab in complex with BG505 DS-SOSIP.664
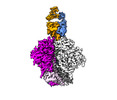
EMDB-29288: 
Cryo-EM Structure of PGT145 DU303 Fab in complex with BG505 DS-SOSIP.664

PDB-8fk5: 
Cryo-EM Structure of PG9RSH DU011 Fab in complex with BG505 DS-SOSIP.664

PDB-8fl1: 
Cryo-EM Structure of PG9RSH DU025 Fab in complex with BG505 DS-SOSIP.664

PDB-8flw: 
Cryo-EM Structure of PGT145 DU303 Fab in complex with BG505 DS-SOSIP.664

EMDB-34276: 
Cryo-EM structure of CB2-G protein complex

EMDB-34277: 
Cryo-EM structure of CP-CB2-G protein complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
