-検索条件
-検索結果
検索 (著者・登録者: pei & w & lian)の結果295件中、1から50件目までを表示しています

EMDB-34928: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 S-trimer in complex with fab L4.65 and L5.34

EMDB-34940: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4 S-trimer in complex with fab L4.65 and L5.34

EMDB-34945: 
Cryo-EM structure of SARS-CoV-2 Omicron Prototype S-trimer in complex with fab L4.65 and L5.34

PDB-8hp9: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 S-trimer in complex with fab L4.65 and L5.34

PDB-8hpq: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4 S-trimer in complex with fab L4.65 and L5.34

PDB-8hpv: 
Cryo-EM structure of SARS-CoV-2 Omicron Prototype S-trimer in complex with fab L4.65 and L5.34

EMDB-34931: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with fab L4.65 and L5.34

EMDB-34944: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4 RBD in complex with fab L4.65 and L5.34

EMDB-34946: 
Cryo-EM structure of SARS-CoV-2 Omicron Prototype RBD in complex with fab L4.65 and L5.34

PDB-8hpf: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with fab L4.65 and L5.34

PDB-8hpu: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4 RBD in complex with fab L4.65 and L5.34

PDB-8hq7: 
Cryo-EM structure of SARS-CoV-2 Omicron Prototype RBD in complex with fab L4.65 and L5.34
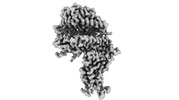
EMDB-33841: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.12.1 RBD in complex with human ACE2 (local refinement)
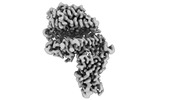
EMDB-33870: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with human ACE2 (local refinement)
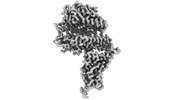
EMDB-34120: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with golden hamster ACE2 (local refinement)

EMDB-34138: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with mouse ACE2 (local refinement)

EMDB-34217: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with rat ACE2 (local refinement)

EMDB-34409: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4/5 RBD in complex with human ACE2 (local refinement)

EMDB-34494: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with human ACE2 (three-RBD-up conformation)

EMDB-34498: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2.12.1 spike trimer in complex with human ACE2 (three-RBD-up conformation)

EMDB-34499: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike protein in complex with mouse ACE2

EMDB-34506: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with rat ACE2

EMDB-34509: 
Cryo-EM map of SARS-CoV-2 Omicron BA.4/5 (N658S) spike trimer in complex with human ACE2 (three-RBD-up conformation)
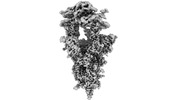
EMDB-34510: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with golden hamster ACE2

PDB-7yhw: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.12.1 RBD in complex with human ACE2 (local refinement)

PDB-7yj3: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with human ACE2 (local refinement)

PDB-7yv8: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with golden hamster ACE2 (local refinement)

PDB-7yvu: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with mouse ACE2 (local refinement)

PDB-8gry: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with rat ACE2 (local refinement)

PDB-8h06: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4/5 RBD in complex with human ACE2 (local refinement)

EMDB-34202: 
AK-42 inhibitor binding human ClC-2 TMD

PDB-8gqu: 
AK-42 inhibitor binding human ClC-2 TMD

EMDB-34710: 
Cryo-EM structure of WeiTsing

PDB-8hf2: 
Cryo-EM structure of WeiTsing

EMDB-15763: 
Untreatment WT HIV-1 immature gag

EMDB-15764: 
PDDC treatment HIV immature Gag
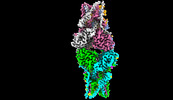
EMDB-35503: 
Cryo-EM structure of Stimulator of interferon genes

EMDB-35504: 
Structure of Stimulator of interferon genes/ligand complex

PDB-8ik0: 
Cryo-EM structure of Stimulator of interferon genes

PDB-8ik3: 
Structure of Stimulator of interferon genes/ligand complex
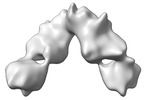
EMDB-33225: 
Cryo-EM structure of SARS-CoV-2 prototype RBD-dimer bound to CB6 Fab
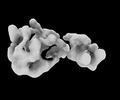
EMDB-35767: 
Structure of the Mex67-Mtr2-3 heterodimer

EMDB-34638: 
Structure of the Mex67-Mtr2-1 heterodimer
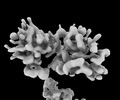
EMDB-34640: 
Structure of Mex67-Mtr2-2 heterodimer
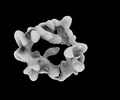
EMDB-34641: 
Structure of Crm1-RanGTP complex

EMDB-34725: 
NPC-trapped pre-60S particle
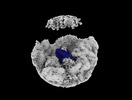
EMDB-35812: 
Bud20 interacts with CFNC

PDB-8hbn: 
Structure of the Mex67-Mtr2-1 heterodimer

PDB-8hfr: 
NPC-trapped pre-60S particle

EMDB-33633: 
Cryo-EM structure of C5a-bound C5aR1 in complex with Gi protein
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
