-検索条件
-検索結果
検索 (著者・登録者: bhella & d)の結果52件中、1から50件目までを表示しています

EMDB-16364: 
The lipid linked oligosaccharide polymerase Wzy and its regulating co-polymerase Wzz form a complex in vivo and in vitro

PDB-8c0e: 
The lipid linked oligosaccharide polymerase Wzy and its regulating co-polymerase Wzz form a complex in vivo and in vitro

EMDB-16572: 
1.85 angstrom resolution cryo-EM reconstruction of tobacco mosaic virus, imaged on a JEOL CryoARM 300 with Direct Electron Apollo camera.

EMDB-13228: 
Cryo-electron tomogram of a hybrid virus particle generated by coinfection of human Influenza A virus and Respiratory Syncytial virus on lung cells.

EMDB-13229: 
Cryo-electron tomogram of a pseudotype virus particle generated during coinfection of Influenza A virus and Respiratory Syncytial virus in lung cells.

EMDB-13856: 
Tomogram of respiratory syncytial virus filamentous virion

EMDB-13855: 
Structure of respiratory syncytial virus matrix layer

EMDB-12093: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12094: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12096: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12097: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12098: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12099: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12100: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12101: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12102: 
Higher-order structures of the foot-and-mouth disease virus RNA-dependent RNA polymerase required for dynamic inter-molecular interactions involved in viral genome replication

EMDB-12194: 
The cryo-EM structure of vesivirus 2117, an adventitious agent and possible cause of haemorrhagic gastroenteritis in dogs.

PDB-7bjp: 
The cryo-EM structure of vesivirus 2117, an adventitious agent and possible cause of haemorrhagic gastroenteritis in dogs.

EMDB-10736: 
Lumazine Synthase

EMDB-4549: 
Cryo-EM structure of the GII.4 CHDC-1974 VLP with T=4 icosahedral symmetry

EMDB-4550: 
Cryo-EM structure of human norovirus GII.4 NSW-2012 VLP with T=4 icosahedral symmetry

EMDB-0054: 
Feline Calicivirus Strain F9

EMDB-0056: 
Feline Calicivirus Strain F9 bound to a soluble ectodomain fragment of feline junctional adhesion molecule A - leading to assembly of a portal structure at a unique three-fold axis.

PDB-6gsh: 
Feline Calicivirus Strain F9

PDB-6gsi: 
Feline Calicivirus Strain F9 bound to a soluble ectodomain fragment of feline junctional adhesion molecule A - leading to assembly of a portal structure at a unique three-fold axis.

EMDB-0130: 
Structure of the Macrobrachium rosenbergii Nodavirus

EMDB-0129: 
Structure of the Macrobrachium rosenbergii Nodavirus

PDB-6h2b: 
Structure of the Macrobrachium rosenbergii Nodavirus

EMDB-4347: 
Structure of the herpes-simplex virus portal-vertex

EMDB-3655: 
Cryo-Electron Microscopy Structure of the Macrobrachium rosenbergii Nodavirus Capsid at 7 Angstroms Resolution

PDB-4bkk: 
The Respiratory Syncytial Virus nucleoprotein-RNA complex forms a left-handed helical nucleocapsid.
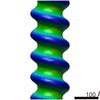
EMDB-2369: 
The Respiratory Syncytial Virus nucleoprotein-RNA complex forms a left-handed helical nucleocapsid.

EMDB-2370: 
The Respiratory Syncytial Virus nucleoprotein-RNA complex forms a left-handed helical nucleocapsid.

EMDB-5452: 
A Tail-like Assembly at the Portal Vertex in Intact Herpes Simplex Type-1 Virions

EMDB-5453: 
A Tail-like Assembly at the Portal Vertex in Intact Herpes Simplex Type-1 Virions

EMDB-1942: 
Feline Calicivirus strain F9

EMDB-1943: 
Feline Calicivirus strain F9 decorated with Junctional Adhesion Molecule A

EMDB-1944: 
Feline Calicivirus strain F9 decorated with Junctional Adhesion Molecule A

EMDB-1945: 
Feline Calicivirus strain F9 decorated with Junctional Adhesion Molecule A

EMDB-1946: 
Feline Calicivirus strain F9 decorated with Junctional Adhesion Molecule A

EMDB-1947: 
Feline Calicivirus strain F9 decorated with Junctional Adhesion Molecule A

EMDB-1948: 
Feline Calicivirus strain F9 decorated with Junctional Adhesion Molecule A

EMDB-1658: 
Solution structure and characterisation of the human pyruvate dehydrogenase complex core assembly

EMDB-1659: 
Solution structure and characterisation of the human pyruvate dehydrogenase complex core assembly

EMDB-1622: 
Helical reconstruction of Respiratory Syncytial Virus N-RNA helices

EMDB-1464: 
Adenovirus serotype 5 hexon mediates liver gene transfer.

PDB-2c8i: 
Complex Of Echovirus Type 12 With Domains 1, 2, 3 and 4 Of Its Receptor Decay Accelerating Factor (Cd55) By Cryo Electron Microscopy At 16 A

EMDB-1182: 
Structural and functional insights into the interaction of echoviruses and decay-accelerating factor.

EMDB-1183: 
Structural and functional insights into the interaction of echoviruses and decay-accelerating factor.

PDB-1upn: 
COMPLEX OF ECHOVIRUS TYPE 12 WITH DOMAINS 3 AND 4 OF ITS RECEPTOR DECAY ACCELERATING FACTOR (CD55) BY CRYO ELECTRON MICROSCOPY AT 16 A
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
