+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8839 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The Hsp90 Co-chaperon R2TP Forms a Hexameric Platform | ||||||||||||||||||
 マップデータ マップデータ | Rvb1 and Rvb2 complexed with Tah1 and Pih1. Map with bfactor compensation applied. | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 生物種 |   Saccharomyces cerevisiae S288c (パン酵母) Saccharomyces cerevisiae S288c (パン酵母) | ||||||||||||||||||
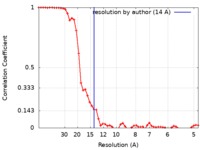
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 14.0 Å クライオ電子顕微鏡法 / 解像度: 14.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Tian S / Yu G / He H / Liu P / Marshall A / Demeler B / Stagg S / Li H | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2017 ジャーナル: Structure / 年: 2017タイトル: Pih1p-Tah1p Puts a Lid on Hexameric AAA+ ATPases Rvb1/2p. 著者: Shaoxiong Tian / Ge Yu / Huan He / Yu Zhao / Peilu Liu / Alan G Marshall / Borries Demeler / Scott M Stagg / Hong Li /  要旨: The Saccharomyces cerevisiae (Sc) R2TP complex affords an Hsp90-mediated and nucleotide-driven chaperone activity to proteins of small ribonucleoprotein particles (snoRNPs). The current lack of ...The Saccharomyces cerevisiae (Sc) R2TP complex affords an Hsp90-mediated and nucleotide-driven chaperone activity to proteins of small ribonucleoprotein particles (snoRNPs). The current lack of structural information on the ScR2TP complex, however, prevents a mechanistic understanding of this biological process. We characterized the structure of the ScR2TP complex made up of two AAA+ ATPases, Rvb1/2p, and two Hsp90 binding proteins, Tah1p and Pih1p, and its interaction with the snoRNP protein Nop58p by a combination of analytical ultracentrifugation, isothermal titration calorimetry, chemical crosslinking, hydrogen-deuterium exchange, and cryoelectron microscopy methods. We find that Pih1p-Tah1p interacts with Rvb1/2p cooperatively through the nucleotide-sensitive domain of Rvb1/2p. Nop58p further binds Pih1p-Tahp1 on top of the dome-shaped R2TP. Consequently, nucleotide binding releases Pih1p-Tah1p from Rvb1/2p, which offers a mechanism for nucleotide-driven binding and release of snoRNP intermediates. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8839.map.gz emd_8839.map.gz | 6.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8839-v30.xml emd-8839-v30.xml emd-8839.xml emd-8839.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8839_fsc.xml emd_8839_fsc.xml | 6.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8839.png emd_8839.png | 122.1 KB | ||
| その他 |  emd_8839_half_map_1.map.gz emd_8839_half_map_1.map.gz emd_8839_half_map_2.map.gz emd_8839_half_map_2.map.gz | 874.4 KB 3.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8839 http://ftp.pdbj.org/pub/emdb/structures/EMD-8839 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8839 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8839 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8839.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8839.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Rvb1 and Rvb2 complexed with Tah1 and Pih1. Map with bfactor compensation applied. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.43 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Rvb1 and Rvb2 complexed with Tah1 and Pih1. Halfmap1
| ファイル | emd_8839_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Rvb1 and Rvb2 complexed with Tah1 and Pih1. Halfmap1 | ||||||||||||
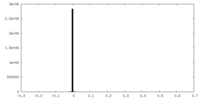
| 投影像・断面図 |
| ||||||||||||
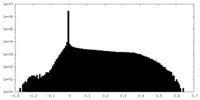
| 密度ヒストグラム |
-ハーフマップ: Rvb1 and Rvb2 complexed with Tah1 and Pih1. Halfmap2
| ファイル | emd_8839_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Rvb1 and Rvb2 complexed with Tah1 and Pih1. Halfmap2 | ||||||||||||
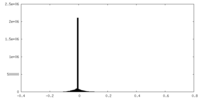
| 投影像・断面図 |
| ||||||||||||
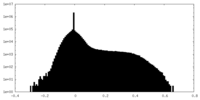
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Rvb1 and Rvb2 together with Tah1 and Pih1
| 全体 | 名称: Complex of Rvb1 and Rvb2 together with Tah1 and Pih1 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Rvb1 and Rvb2 together with Tah1 and Pih1
| 超分子 | 名称: Complex of Rvb1 and Rvb2 together with Tah1 and Pih1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288c (パン酵母) Saccharomyces cerevisiae S288c (パン酵母) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 358 KDa |
-分子 #1: Rvb1p
| 分子 | 名称: Rvb1p / タイプ: other / ID: 1 / 分類: other |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288c (パン酵母) Saccharomyces cerevisiae S288c (パン酵母) |
| 配列 | 文字列: MVAISEVKEN PGVNSSNSGA VTRTAAHTHI KGLGLDESGV AKRVEGGFVG QIEAREACGV IVDLIKAKKM SGRAILLAG GPSTGKTALA LAISQELGPK VPFCPLVGSE LYSVEVKKTE TLMENFRRAI GLRIKETKEV Y EGEVTELT PEDAENPLGG YGKTISHVIV ...文字列: MVAISEVKEN PGVNSSNSGA VTRTAAHTHI KGLGLDESGV AKRVEGGFVG QIEAREACGV IVDLIKAKKM SGRAILLAG GPSTGKTALA LAISQELGPK VPFCPLVGSE LYSVEVKKTE TLMENFRRAI GLRIKETKEV Y EGEVTELT PEDAENPLGG YGKTISHVIV GLKSAKGTKT LRLDPTIYES IQREKVSIGD VIYIEANTGA VK RVGRSDA YATEFDLETE EYVPLPKGEV HKKKEIVQDV TLHDLDVANA RPQGGQDVIS MMGQLLKPKK TEI TEKLRQ EVNKVVAKYI DQGVAELIPG VLFIDEVNML DIEIFTYLNK ALESNIAPVV VLASNRGMTT VRGT EDVIS PHGVPPDLID RLLIVRTLPY DKDEIRTIIE RRATVERLQV ESSALDLLAT MGTETSLRYA LQLLA PCGI LAQTSNRKEI VVNDVNEAKL LFLDAKRSTK ILETSANYL |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #2: Rvb2p
| 分子 | 名称: Rvb2p / タイプ: other / ID: 2 / 分類: other |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288c (パン酵母) Saccharomyces cerevisiae S288c (パン酵母) |
| 配列 | 文字列: MSIQTSDPNE TSDLKSLSLI AAHSHITGLG LDENLQPRPT SEGMVGQLQA RRAAGVILKM VQNGTIAGRA VLVAGPPST GKTALAMGVS QSLGKDVPFT AIAGSEIFSL ELSKTEALTQ AFRKSIGIKI KEETELIEGE V VEIQIDRS ITGGHKQGKL TIKTTDMETI ...文字列: MSIQTSDPNE TSDLKSLSLI AAHSHITGLG LDENLQPRPT SEGMVGQLQA RRAAGVILKM VQNGTIAGRA VLVAGPPST GKTALAMGVS QSLGKDVPFT AIAGSEIFSL ELSKTEALTQ AFRKSIGIKI KEETELIEGE V VEIQIDRS ITGGHKQGKL TIKTTDMETI YELGNKMIDG LTKEKVLAGD VISIDKASGK ITKLGRSFAR SR DYDAMGA DTRFVQCPEG ELQKRKTVVH TVSLHEIDVI NSRTQGFLAL FTGDTGEIRS EVRDQINTKV AEW KEEGKA EIVPGVLFID EVHMLDIECF SFINRALEDE FAPIVMMATN RGVSKTRGTN YKSPHGLPLD LLDR SIIIT TKSYNEQEIK TILSIRAQEE EVELSSDALD LLTKTGVETS LRYSSNLISV AQQIAMKRKN NTVEV EDVK RAYLLFLDSA RSVKYVQENE SQYIDDQGNV QISIAKSADP DAMDTTE |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #3: Pih1p
| 分子 | 名称: Pih1p / タイプ: other / ID: 3 / 分類: other |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288c (パン酵母) Saccharomyces cerevisiae S288c (パン酵母) |
| 配列 | 文字列: MADFLLRPIK QRHRNEDKYV SVDAADGSVS KIEPIADFVI KTKLLSANGP EKLQDGRKVF INVCHSPLVP KPEVDFNAR IVFPLIIQNE WEIPIITSCY RMDHDKKGQE CYVWDCCINS DCSRWICDDI QLREILVEWC L ESCEIRDS VVLCRDRIAF PKMKKKGAEL ...文字列: MADFLLRPIK QRHRNEDKYV SVDAADGSVS KIEPIADFVI KTKLLSANGP EKLQDGRKVF INVCHSPLVP KPEVDFNAR IVFPLIIQNE WEIPIITSCY RMDHDKKGQE CYVWDCCINS DCSRWICDDI QLREILVEWC L ESCEIRDS VVLCRDRIAF PKMKKKGAEL PALEVLNDEL HQDYKAKMHK IIEEEAGDPM SILRGRNDDG DD NNDPDDG TLPPLFPIEN KISGAKIEEI DKNEIAHRNL KQAPAPAPAP HEQQEDVPEY EVKMKRFKGA AYK LRILIE NKAPNSKPDR FSPSYNFAEN ILYINGKLSI PLPRDIVVNA ADIKIFHIRK ERTLYIYI |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #4: Tah1p
| 分子 | 名称: Tah1p / タイプ: other / ID: 4 / 分類: other |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288c (パン酵母) Saccharomyces cerevisiae S288c (パン酵母) |
| 配列 | 文字列: MSQFEKQKEQ GNSLFKQGLY REAVHCYDQL ITAQPQNPVG YSNKAMALIK LGEYTQAIQM CQQGLRYTST AEHVAIRSK LQYRLELAQG AVGSVQIPVV EVDELPEGYD RS |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): -3.0 µm / 最小 デフォーカス(補正後): -1.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 29000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 83.0 K / 最高: 93.0 K |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 検出モード: INTEGRATING / 実像数: 1396 / 平均電子線量: 58.68 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X