+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8796 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Katanin hexamer in the ring conformation | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | microtubule cytoskeleton / microtubule severing protein /  AAA ATPase / p60 / AAA ATPase / p60 /  MOTOR PROTEIN (モータータンパク質) MOTOR PROTEIN (モータータンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of meiotic spindle elongation /  MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly / MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly /  katanin complex / meiotic spindle pole / katanin complex / meiotic spindle pole /  microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division ...negative regulation of meiotic spindle elongation / microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division ...negative regulation of meiotic spindle elongation /  MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly / MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly /  katanin complex / meiotic spindle pole / katanin complex / meiotic spindle pole /  microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division / meiotic spindle organization / microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division / meiotic spindle organization /  紡錘体 / embryo development ending in birth or egg hatching / microtubule depolymerization / 紡錘体 / embryo development ending in birth or egg hatching / microtubule depolymerization /  isomerase activity / spindle / isomerase activity / spindle /  紡錘体 / microtubule cytoskeleton / 紡錘体 / microtubule cytoskeleton /  microtubule binding / microtubule binding /  protein phosphatase binding / protein phosphatase binding /  微小管 / molecular adaptor activity / 微小管 / molecular adaptor activity /  細胞分裂 / 細胞分裂 /  中心体 / 中心体 /  クロマチン / クロマチン /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) | |||||||||
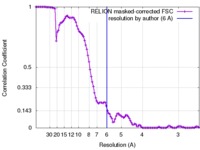
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.0 Å クライオ電子顕微鏡法 / 解像度: 6.0 Å | |||||||||
 データ登録者 データ登録者 | Zehr EA / Szyk A | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2017 ジャーナル: Nat Struct Mol Biol / 年: 2017タイトル: Katanin spiral and ring structures shed light on power stroke for microtubule severing. 著者: Elena Zehr / Agnieszka Szyk / Grzegorz Piszczek / Ewa Szczesna / Xiaobing Zuo / Antonina Roll-Mecak /  要旨: Microtubule-severing enzymes katanin, spastin and fidgetin are AAA ATPases important for the biogenesis and maintenance of complex microtubule arrays in axons, spindles and cilia. Because of a lack ...Microtubule-severing enzymes katanin, spastin and fidgetin are AAA ATPases important for the biogenesis and maintenance of complex microtubule arrays in axons, spindles and cilia. Because of a lack of known 3D structures for these enzymes, their mechanism of action has remained poorly understood. Here we report the X-ray crystal structure of the monomeric AAA katanin module from Caenorhabditis elegans and cryo-EM reconstructions of the hexamer in two conformations. The structures reveal an unexpected asymmetric arrangement of the AAA domains mediated by structural elements unique to microtubule-severing enzymes and critical for their function. The reconstructions show that katanin cycles between open spiral and closed ring conformations, depending on the ATP occupancy of a gating protomer that tenses or relaxes interprotomer interfaces. Cycling of the hexamer between these conformations would provide the power stroke for microtubule severing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8796.map.gz emd_8796.map.gz | 5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8796-v30.xml emd-8796-v30.xml emd-8796.xml emd-8796.xml | 13 KB 13 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8796_fsc.xml emd_8796_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8796.png emd_8796.png | 111.3 KB | ||
| Filedesc metadata |  emd-8796.cif.gz emd-8796.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8796 http://ftp.pdbj.org/pub/emdb/structures/EMD-8796 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8796 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8796 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8796.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8796.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : katanin
| 全体 | 名称: katanin カタニン カタニン |
|---|---|
| 要素 |
|
-超分子 #1: katanin
| 超分子 | 名称: katanin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
| 分子量 | 理論値: 310 KDa |
-分子 #1: Meiotic spindle formation protein mei-1
| 分子 | 名称: Meiotic spindle formation protein mei-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: ec: 3.6.4.3 |
|---|---|
| 由来(天然) | 生物種:   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
| 分子量 | 理論値: 51.802359 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MNGDVQSVIR GYLERAQVAK TMSDAGRWNE AGDLLRQLMT DVKSCKISAS NRDEHDARNT FLRALEANLK LVQQNVRDED DLHEAMTRQ SGSPEPPADP DVWSKPSPPL PSSSKFGATK KGVGAAGPRP REISKSTSSM STNPADVKPA NPTQGILPQN S AGDSFDAS ...文字列: MNGDVQSVIR GYLERAQVAK TMSDAGRWNE AGDLLRQLMT DVKSCKISAS NRDEHDARNT FLRALEANLK LVQQNVRDED DLHEAMTRQ SGSPEPPADP DVWSKPSPPL PSSSKFGATK KGVGAAGPRP REISKSTSSM STNPADVKPA NPTQGILPQN S AGDSFDAS AYDAYIVQAV RGTMATNTEN TMSLDDIIGM HDVKQVLHEA VTLPLLVPEF FQGLRSPWKA MVLAGPPGTG KT LIARAIA SESSSTFFTV SSTDLSSKWR GDSEKIVRLL FELARFYAPS IIFIDQIDTL GGQRGNSGEH EASRRVKSEF LVQ MDGSQN KFDSRRVFVL AATNIPWELD EALRRRFEKR IFIPLPDIDA RKKLIEKSME GTPKSDEINY DDLAARTEGF SGAD VVSLC RTAAINVLRR YDTKSLRGGE LTAAMESLKA ELVRNIDFEA ALQAVSPSAG PDTMLKCKEW CDSFGAM UniProtKB: Meiotic spindle formation protein mei-1 |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 5 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 6 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 298 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3837 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 1 / 実像数: 2100 / 平均露光時間: 17.5 sec. / 平均電子線量: 51.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー