+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6803 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
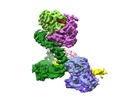
| タイトル | Cryo-EM structure of Human DNA-PK Holoenzyme | |||||||||||||||
 マップデータ マップデータ | None | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Cryo-EM structure / DNA-PK / DNAPKcs /  activation (活性化) / activation (活性化) /  NHEJ (非相同末端結合) / NHEJ (非相同末端結合) /  DNA BINDING PROTEIN (DNA結合タンパク質) DNA BINDING PROTEIN (DNA結合タンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Ku70:Ku80 complex / negative regulation of t-circle formation / positive regulation of platelet formation /  DNA end binding / T cell receptor V(D)J recombination / pro-B cell differentiation / small-subunit processome assembly / positive regulation of lymphocyte differentiation / DNA-dependent protein kinase activity / histone H2AXS139 kinase activity ...Ku70:Ku80 complex / negative regulation of t-circle formation / positive regulation of platelet formation / DNA end binding / T cell receptor V(D)J recombination / pro-B cell differentiation / small-subunit processome assembly / positive regulation of lymphocyte differentiation / DNA-dependent protein kinase activity / histone H2AXS139 kinase activity ...Ku70:Ku80 complex / negative regulation of t-circle formation / positive regulation of platelet formation /  DNA end binding / T cell receptor V(D)J recombination / pro-B cell differentiation / small-subunit processome assembly / positive regulation of lymphocyte differentiation / DNA-dependent protein kinase activity / histone H2AXS139 kinase activity / DNA-dependent protein kinase complex / immature B cell differentiation / DNA-dependent protein kinase-DNA ligase 4 complex / cellular response to X-ray / immunoglobulin V(D)J recombination / DNA end binding / T cell receptor V(D)J recombination / pro-B cell differentiation / small-subunit processome assembly / positive regulation of lymphocyte differentiation / DNA-dependent protein kinase activity / histone H2AXS139 kinase activity / DNA-dependent protein kinase complex / immature B cell differentiation / DNA-dependent protein kinase-DNA ligase 4 complex / cellular response to X-ray / immunoglobulin V(D)J recombination /  nonhomologous end joining complex / nonhomologous end joining complex /  DNA ligation / nuclear telomere cap complex / regulation of smooth muscle cell proliferation / double-strand break repair via alternative nonhomologous end joining / Cytosolic sensors of pathogen-associated DNA / double-strand break repair via classical nonhomologous end joining / regulation of epithelial cell proliferation / IRF3-mediated induction of type I IFN / telomere capping / DNA ligation / nuclear telomere cap complex / regulation of smooth muscle cell proliferation / double-strand break repair via alternative nonhomologous end joining / Cytosolic sensors of pathogen-associated DNA / double-strand break repair via classical nonhomologous end joining / regulation of epithelial cell proliferation / IRF3-mediated induction of type I IFN / telomere capping /  相同組換え / 相同組換え /  regulation of telomere maintenance / U3 snoRNA binding / regulation of hematopoietic stem cell differentiation / positive regulation of neurogenesis / protein localization to chromosome, telomeric region / cellular response to fatty acid / hematopoietic stem cell proliferation / cellular hyperosmotic salinity response / maturation of 5.8S rRNA / T cell lineage commitment / negative regulation of cGAS/STING signaling pathway / telomeric DNA binding / positive regulation of catalytic activity / B cell lineage commitment / 2-LTR circle formation / positive regulation of double-strand break repair via nonhomologous end joining / site of DNA damage / regulation of telomere maintenance / U3 snoRNA binding / regulation of hematopoietic stem cell differentiation / positive regulation of neurogenesis / protein localization to chromosome, telomeric region / cellular response to fatty acid / hematopoietic stem cell proliferation / cellular hyperosmotic salinity response / maturation of 5.8S rRNA / T cell lineage commitment / negative regulation of cGAS/STING signaling pathway / telomeric DNA binding / positive regulation of catalytic activity / B cell lineage commitment / 2-LTR circle formation / positive regulation of double-strand break repair via nonhomologous end joining / site of DNA damage /  付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; その他の炭素-酸素リアーゼ / 付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; その他の炭素-酸素リアーゼ /  enzyme activator activity / 5'-deoxyribose-5-phosphate lyase activity / hematopoietic stem cell differentiation / positive regulation of protein kinase activity / ectopic germ cell programmed cell death / ATP-dependent activity, acting on DNA / enzyme activator activity / 5'-deoxyribose-5-phosphate lyase activity / hematopoietic stem cell differentiation / positive regulation of protein kinase activity / ectopic germ cell programmed cell death / ATP-dependent activity, acting on DNA /  somitogenesis / positive regulation of telomerase activity / mitotic G1 DNA damage checkpoint signaling / positive regulation of telomere maintenance via telomerase / somitogenesis / positive regulation of telomerase activity / mitotic G1 DNA damage checkpoint signaling / positive regulation of telomere maintenance via telomerase /  DNA helicase activity / DNA helicase activity /  telomere maintenance / activation of innate immune response / cellular response to leukemia inhibitory factor / small-subunit processome / telomere maintenance / activation of innate immune response / cellular response to leukemia inhibitory factor / small-subunit processome /  神経発生 / 神経発生 /  cyclin binding / positive regulation of translation / negative regulation of protein phosphorylation / positive regulation of erythrocyte differentiation / cyclin binding / positive regulation of translation / negative regulation of protein phosphorylation / positive regulation of erythrocyte differentiation /  デオキシリボ核酸 / response to gamma radiation / Nonhomologous End-Joining (NHEJ) / デオキシリボ核酸 / response to gamma radiation / Nonhomologous End-Joining (NHEJ) /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 /  brain development / peptidyl-threonine phosphorylation / protein modification process / protein destabilization / cellular response to gamma radiation / brain development / peptidyl-threonine phosphorylation / protein modification process / protein destabilization / cellular response to gamma radiation /  regulation of circadian rhythm / double-strand break repair via nonhomologous end joining / cellular response to insulin stimulus / rhythmic process / intrinsic apoptotic signaling pathway in response to DNA damage / double-strand break repair / E3 ubiquitin ligases ubiquitinate target proteins / T cell differentiation in thymus / regulation of circadian rhythm / double-strand break repair via nonhomologous end joining / cellular response to insulin stimulus / rhythmic process / intrinsic apoptotic signaling pathway in response to DNA damage / double-strand break repair / E3 ubiquitin ligases ubiquitinate target proteins / T cell differentiation in thymus /  heart development / heart development /  double-stranded DNA binding / double-stranded DNA binding /  scaffold protein binding / peptidyl-serine phosphorylation / secretory granule lumen / DNA recombination / ficolin-1-rich granule lumen / scaffold protein binding / peptidyl-serine phosphorylation / secretory granule lumen / DNA recombination / ficolin-1-rich granule lumen /  transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding / damaged DNA binding / transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding / damaged DNA binding /  chromosome, telomeric region / transcription cis-regulatory region binding / chromosome, telomeric region / transcription cis-regulatory region binding /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  protein kinase activity / response to xenobiotic stimulus / protein kinase activity / response to xenobiotic stimulus /  ribonucleoprotein complex / positive regulation of apoptotic process / protein domain specific binding / ribonucleoprotein complex / positive regulation of apoptotic process / protein domain specific binding /  protein phosphorylation protein phosphorylation類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
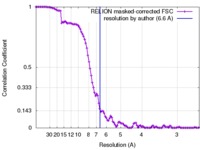
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.6 Å クライオ電子顕微鏡法 / 解像度: 6.6 Å | |||||||||||||||
 データ登録者 データ登録者 | Yin X / Liu M | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2017 ジャーナル: Cell Res / 年: 2017タイトル: Cryo-EM structure of human DNA-PK holoenzyme. 著者: Xiaotong Yin / Mengjie Liu / Yuan Tian / Jiawei Wang / Yanhui Xu /  要旨: DNA-dependent protein kinase (DNA-PK) is a serine/threonine protein kinase complex composed of a catalytic subunit (DNA-PKcs) and KU70/80 heterodimer bound to DNA. DNA-PK holoenzyme plays a critical ...DNA-dependent protein kinase (DNA-PK) is a serine/threonine protein kinase complex composed of a catalytic subunit (DNA-PKcs) and KU70/80 heterodimer bound to DNA. DNA-PK holoenzyme plays a critical role in non-homologous end joining (NHEJ), the major DNA repair pathway. Here, we determined cryo-electron microscopy structure of human DNA-PK holoenzyme at 6.6 Å resolution. In the complex structure, DNA-PKcs, KU70, KU80 and DNA duplex form a 650-kDa heterotetramer with 1:1:1:1 stoichiometry. The N-terminal α-solenoid (∼2 800 residues) of DNA-PKcs adopts a double-ring fold and connects the catalytic core domain of DNA-PKcs and KU70/80-DNA. DNA-PKcs and KU70/80 together form a DNA-binding tunnel, which cradles ∼30-bp DNA and prevents sliding inward of DNA-PKcs along with DNA duplex, suggesting a mechanism by which the broken DNA end is protected from unnecessary processing. Structural and biochemical analyses indicate that KU70/80 and DNA coordinately induce conformational changes of DNA-PKcs and allosterically stimulate its kinase activity. We propose a model for activation of DNA-PKcs in which allosteric signals are generated upon DNA-PK holoenzyme formation and transmitted to the kinase domain through N-terminal HEAT repeats and FAT domain of DNA-PKcs. Our studies suggest a mechanism for recognition and protection of broken DNA ends and provide a structural basis for understanding the activation of DNA-PKcs and DNA-PK-mediated NHEJ pathway. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6803.map.gz emd_6803.map.gz | 6.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6803-v30.xml emd-6803-v30.xml emd-6803.xml emd-6803.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6803_fsc.xml emd_6803_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6803.png emd_6803.png | 116.9 KB | ||
| Filedesc metadata |  emd-6803.cif.gz emd-6803.cif.gz | 9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6803 http://ftp.pdbj.org/pub/emdb/structures/EMD-6803 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6803 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6803 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6803.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6803.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PRKDC-Helix
| 全体 | 名称: PRKDC-Helix |
|---|---|
| 要素 |
|
-超分子 #1: PRKDC-Helix
| 超分子 | 名称: PRKDC-Helix / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: X-ray repair cross-complementing protein 6
| 分子 | 名称: X-ray repair cross-complementing protein 6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号:  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 57.619352 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GRDSLIFLVD ASKAMFESQS EDELTPFDMS IQCIQSVYIS KIISSDRDLL AVVFYGTEKD KNSVNFKNIY VLQELDNPGA KRILELDQF KGQQGQKRFQ DMMGHGSDYS LSEVLWVCAN LFSDVQFKMS HKRIMLFTNE DNPHGNDSAK ASRARTKAGD L RDTGIFLD ...文字列: GRDSLIFLVD ASKAMFESQS EDELTPFDMS IQCIQSVYIS KIISSDRDLL AVVFYGTEKD KNSVNFKNIY VLQELDNPGA KRILELDQF KGQQGQKRFQ DMMGHGSDYS LSEVLWVCAN LFSDVQFKMS HKRIMLFTNE DNPHGNDSAK ASRARTKAGD L RDTGIFLD LMHLKKPGGF DISLFYRDII SIAEDEDLRV HFEESSKLED LLRKVRAKET RKRALSRLKL KLNKDIVISV GI YNLVQKA LKPPPIKLYR ETNEPVKTKT RTFNTSTGGL LLPSDTKRSQ IYGSRQIILE KEETEELKRF DDPGLMLMGF KPL VLLKKH HYLRPSLFVY PEESLVIGSS TLFSALLIKC LEKEVAALCR YTPRRNIPPY FVALVPQEEE LDDQKIQVTP PGFQ LVFLP FADDKRKMPF TEKIMATPEQ VGKMKAIVEK LRFTYRSDSF ENPVLQQHFR NLEALALDLM EPEQAVDLTL PKVEA MNKR LGSLVDEFKE LVYPPDY UniProtKB: X-ray repair cross-complementing protein 6 |
-分子 #2: X-ray repair cross-complementing protein 5
| 分子 | 名称: X-ray repair cross-complementing protein 5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号:  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 60.972969 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: NKAAVVLCMD VGFTMSNSIP GIESPFEQAK KVITMFVQRQ VFAENKDEIA LVLFGTDGTD NPLSGGDQYQ NITVHRHLML PDFDLLEDI ESKIQPGSQQ ADFLDALIVS MDVIQHETIG KKFEKRHIEI FTDLSSRFSK SQLDIIIHSL KKCDISLQFF L PFSLGKED ...文字列: NKAAVVLCMD VGFTMSNSIP GIESPFEQAK KVITMFVQRQ VFAENKDEIA LVLFGTDGTD NPLSGGDQYQ NITVHRHLML PDFDLLEDI ESKIQPGSQQ ADFLDALIVS MDVIQHETIG KKFEKRHIEI FTDLSSRFSK SQLDIIIHSL KKCDISLQFF L PFSLGKED GSGDRGDGPF RLGGHGPSFP LKGITEQQKE GLEIVKMVMI SLEGEDGLDE IYSFSESLRK LCVFKKIERH SI HWPCRLT IGSNLSIRIA AYKSILQERV KKTWTVVDAK TLKKEDIQKE TVYCLNDDDE TEVLKEDIIQ GFRYGSDIVP FSK VDEEQM KYKSEGKCFS VLGFCKSSQV QRRFFMGNQV LKVFAARDDE AAAVALSSLI HALDDLDMVA IVRYAYDKRA NPQV GVAFP HIKHNYECLV YVQLPFMEDL RQYMFSSLKN SKKYAPTEAQ LNAVDALIDS MSLAKKDEKT DTLEDLFPTT KIPNP RFQR LFQCLLHRAL HPREPLPPIQ QHIWNMLNPP AEVTTKSQIP LSKIKTLFPL IE UniProtKB: X-ray repair cross-complementing protein 5 |
-分子 #3: PRKDC-Helix
| 分子 | 名称: PRKDC-Helix / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.084182 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AAAAAAAAAA AAAAA |
-分子 #6: DNA-dependent protein kinase catalytic subunit
| 分子 | 名称: DNA-dependent protein kinase catalytic subunit / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO / EC番号:  non-specific serine/threonine protein kinase non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 468.885344 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: CSLLRLQETL SAADRCGAAL AGHQLIRGLG QECVLSSSPA VLALQTSLVF SRDFGLLVFV RKSLNSIEFR ECREEILKFL CIFLEKMGQ KIAPYSVEIK NTCTSVYTKD RAAKCKIPAL DLLIKLLQTF RSSRLMDEFK IGELFSKFYG ELALKKKIPD T VLEKVYEL ...文字列: CSLLRLQETL SAADRCGAAL AGHQLIRGLG QECVLSSSPA VLALQTSLVF SRDFGLLVFV RKSLNSIEFR ECREEILKFL CIFLEKMGQ KIAPYSVEIK NTCTSVYTKD RAAKCKIPAL DLLIKLLQTF RSSRLMDEFK IGELFSKFYG ELALKKKIPD T VLEKVYEL LGLLGEVHPS EMINNAENLF RAFLGELKTQ MTSAVREPKL PVLAGCLKGL SSLLCNFTKS MEEDPQTSRE IF NFVLKAI RPQIDLKRYA VPSAGLRLFA LHASQFSTCL LDNYVSLFEV LLKWCAHTNV ELKKAALSAL ESFLKQVSNM VAK NAEMHK NKLQYFMEQF YGIIRNVDSN NKELSIAIRG YGLFAGPCKV INAKDVDFMY VELIQRCKQM FLTQTDTGDD RVYQ MPSFL QSVASVLLYL DTVPEVYTPV LEHLVVMQID SFPQYSPKMQ LVCCRAIVKV FLALAAKGPV LRNCISTVVH QGLIR ICSK PVVLPKGPES ESEDHRASGE VRTGKWKVPT YKDYVDLFRH LLSSDQMMDS ILADEAFFSV NSSSESLNHL LYDEFV KSV LKIVEKLDLT LEIQTVGEQE NGDEAPGVWM IPTSDPAANL HPAKPKDFSA FINLVEFCRE ILPEKQAEFF EPWVYSF SY ELILQSTRLP LISGFYKLLS ITVRNAKKIK YFEGVSPKSL KHSPEDPEKY SCFALFVKFG KEVAVKMKQY KDELLASC L TFLLSLPHNI IELDVRAYVP ALQMAFKLGL SYTPLAEVGL NALEEWSIYI DRHVMQPYYK DILPCLDGYL KTSALSDET KNNWEVSALS RAAQKGFNKV VLKHLKKTKN LSSNEAISLE EIRIRVVQML GSLGGQINKN LLTVTSSDEM MKSYVAWDRE KRLSFAVPF REMKPVIFLD VFLPRVTELA LTASDRQTKV AACELLHSMV MFMLGKATQM PEGGQGAPPM YQLYKRTFPV L LRLACDVD QVTRQLYEPL VMQLIHWFTN NKKFESQDTV ALLEAILDGI VDPVDSTLRD FCGRCIREFL KWSIKQITPQ QQ EKSPVNT KSLFKRLYSL ALHPNAFKRL GASLAFNNIY REFREEESLV EQFVFEALVI YMESLALAHA DEKSLGTIQQ CCD AIDHLC RIIEKKHVSL NKAKKRRLPR GFPPSASLCL LDLVKWLLAH CGRPQTECRH KSIELFYKFV PLLPGNRSPN LWLK DVLKE EGVSFLINTF EGGGCGQPSG ILAQPTLLYL RGPFSLQATL CWLDLLLAAL ECYNTFIGER TVGALQVLGT EAQSS LLKA VAFFLESIAM HDIIAAEKCF GTGAAGNRTS PQEGERYNYS KCTVVVRIME FTTTLLNTSP EGWKLLKKDL CNTHLM RVL VQTLCEPASI GFNIGDVQVM AHLPDVCVNL MKALKMSPYK DILETHLREK ITAQSIEELC AVNLYGPDAQ VDRSRLA AV VSACKQLHRA GLLHNILPSQ STDLHHSVGT ELLSLVYKGI APGDERQCLP SLDLSCKQLA SGLLELAFAF GGLCERLV S LLLNPAVLST ASLGSSQGSV IHFSHGEYFY SLFSETINTE LLKNLDLAVL ELMQSSVDNT KMVSAVLNGM LDQSFRERA NQKHQGLKLA TTILQHWKKC DSWWAKDSPL ETKMAVLALL AKILQIDSSV SFNTSHGSFP EVFTTYISLL ADTKLDLHLK GQAVTLLPF FTSLTGGSLE ELRRVLEQLI VAHFPMQSRE FPPGTPRFNN YVDCMKKFLD ALELSQSPML LELMTEVLCR E QQHVMEEL FQSSFRRIAR RGSCVTQVGL LESVYEMFRK DDPRLSFTRQ SFVDRSLLTL LWHCSLDALR EFFSTIVVDA ID VLKSRFT KLNESTFDTQ ITKKMGYYKI LDVMYSRLPK DDVHAKESKI NQVFHGSCIT EGNELTKTLI KLCYDAFTEN MAG ENQLLE RRRLYHCAAY NCAISVICCV FNELKFYQGF LFSEKPEKNL LIFENLIDLK RRYNFPVEVE VPMERKKKYI EIRK EAREA ANGDSDGPSY MSSLSYLADS TLSEEMSQFD FSTGVQSYSY SSQDPRPATG RFRRREQRDP TVHDDVLELE MDELN RHEC MAPLTALVKH MHRSLGPPQG EEDSVPRDLP SWMKFLHGKL GNPIVPLNIR LFLAKLVINT EEVFRPYAKH WLSPLL QLA ASENNGGEGI HYMVVEIVAT ILSWTGLATP TGVPKDEVLA NRLLNFLMKH VFHPKRAVFR HNLEIIKTLV ECWKDCL SI PYRLIFEKFS GKDPNSKDNS VGIQLLGIVM ANDLPPYDPQ CGIQSSEYFQ ALVNNMSFVR YKEVYAAAAE VLGLILRY V MERKNILEES LCELVAKQLK QHQNTMEDKF IVCLNKVTKS FPPLADRFMN AVFFLLPKFH GVLKTLCLEV VLCRVEGMT ELYFQLKSKD FVQVMRHRDD ERQKVCLDII YKMMPKLKPV ELRELLNPVV EFVSHPSTTC REQMYNILMW IHDNYRDPES ETDNDSQEI FKLAKDVLIQ GLIDENPGLQ LIIRNFWSHE TRLPSNTLDR LLALNSLYSP KIEVHFLSLA TNFLLEMTSM S PDYPNPMF EHPLSECEFQ EYTIDSDWRF RSTVLTPMFV ETQASQGTLQ TRTQEGSLSA RWPVAGQIRA TQQQHDFTLT QT ADGRSSF DWLTGSSTDP LVDHTSPSSD SLLFAHKRSE RLQRAPLKSV GPDFGKKRLG LPGDEVDNKV KGAAGRTDLL RLR RRFMRD QEKLSLMYAR KGVAEQKREK EIKSELKMKQ DAQVVLYRSY RHGDLPDIQI KHSSLITPLQ AVAQRDPIIA KQLF SSLFS GILKEMDKFK TLSEKNNITQ KLLQDFNRFL NTTFSFFPPF VSCIQDISCQ HAALLSLDPA AVSAGCLASL QQPVG IRLL EEALLRLLPA ELPAKRVRGK ARLPPDVLRW VELAKLYRSI GEYDVLRGIF TSEIGTKQIT QSALLAEARS DYSEAA KQY DEALNKQDWV DGEPTEAEKD FWELASLDCY NHLAEWKSLE YCSTASIDSE NPPDLNKIWS EPFYQETYLP YMIRSKL KL LLQGEADQSL LTFIDKAMHG ELQKAILELH YSQELSLLYL LQDDVDRAKY YIQNGIQSFM QNYSSIDVLL HQSRLTKL Q SVQALTEIQE FISFISKQGN LSSQVPLKRL LNTWTNRYPD AKMDPMNIWD DIITNRCFFL SKIEEKLTPL PEDNSMNVD QDGDPSDRME VQEQEEDISS LIRSCKFSMK MKMIDSARKQ NNFSLAMKLL KELHKESKTR DDWLVSWVQS YCRLSHCRSR SQGCSEQVL TVLKTVSLLD ENNVSSYLSK NILAFRDQNI LLGTTYRIIA NALSSEPACL AEIEEDKARR ILELSGSSSE D SEKVIAGL YQRAFQHLSE AVQAAEEEAQ PPSWSCGPAA GVIDAYMTLA DFCDQQLRKE EENASVIDSA ELQAYPALVV EK MLKALKL NSNEARLKFP RLLQIIERYP EETLSLMTKE ISSVPCWQFI SWISHMVALL DKDQAVAVQH SVEEITDNYP QAI VYPFII SSESYSFKDT STGHKNKEFV ARIKSKLDQG GVIQDFINAL DQLSNPELLF KDWSNDVRAE LAKTPVNKKN IEKM YERMY AALGDPKAPG LGAFRRKFIQ TFGKEFDKHF GKGGSKLLRM KLSDFNDITN MLLLKMNKDS KPPGNLKECS PWMSD FKVE FLRNELEIPG QYDGRGKPLP EYHVRIAGFD ERVTVMASLR RPKRIIIRGH DEREHPFLVK GGEDLRQDQR VEQLFQ VMN GILAQDSACS QRALQLRTYS VVPMTSRLGL IEWLENTVTL KDLLLNTMSQ EEKAAYLSDP RAPPCEYKDW LTKMSGK HD VGAYMLMYKG ANRTETVTSF RKRESKVPAD LLKRAFVRMS TSPEAFLALR SHFASSHALI CISHWILGIG DRHLNNFM V AMETGGVIGI DFGHAFGSAT QFLPVPELMP FRLTRQFINL MLPMKETGLM YSIMVHALRA FRSDPGLLTN TMDVFVKEP SFDWKNFEQK MLKKGGSWIQ EINVAEKNWY PRQKICYAKR KLAGANPAVI TCDELLLGHE KAPAFRDYVA VARGSKDHNI RAQEPESGL SEETQVKCLM DQATDPNILG RTWEGWEPWM UniProtKB: DNA-dependent protein kinase catalytic subunit |
-分子 #4: DNA (34-MER)
| 分子 | 名称: DNA (34-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.502785 KDa |
| 配列 | 文字列: (DT)(DA)(DA)(DA)(DA)(DA)(DC)(DT)(DA)(DT) (DT)(DA)(DT)(DT)(DA)(DT)(DG)(DG)(DT)(DA) (DT)(DT)(DA)(DT)(DG)(DG)(DC)(DC)(DT) (DT)(DG)(DG)(DG)(DC) |
-分子 #5: DNA (36-MER)
| 分子 | 名称: DNA (36-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.042164 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DC)(DT)(DA)(DA)(DT)(DG)(DG) (DC)(DC)(DA)(DT)(DA)(DA)(DT)(DA)(DC)(DC) (DA)(DT)(DA)(DA)(DT)(DA)(DA)(DT)(DA) (DG)(DT)(DT)(DT)(DT)(DT)(DA) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.7 µm / 最小 デフォーカス(公称値): 1.7 µm / 倍率(公称値): 18000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.7 µm / 最小 デフォーカス(公称値): 1.7 µm / 倍率(公称値): 18000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-32 / 実像数: 3496 / 平均露光時間: 8.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー