+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8to2 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
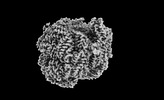
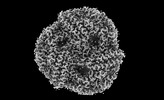
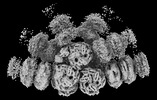
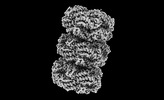
| タイトル | Bottom cylinder of high-resolution phycobilisome quenched by OCP (local refinement) | |||||||||||||||
 要素 要素 |
| |||||||||||||||
 キーワード キーワード |  PHOTOSYNTHESIS (光合成) / PHOTOSYNTHESIS (光合成) /  Complex / light harvesting / Complex / light harvesting /  pigment (顔料) pigment (顔料) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 吸光 / 吸光 /  付加脱離酵素(リアーゼ) / 付加脱離酵素(リアーゼ) /  フィコビリソーム / フィコビリソーム /  chloride ion binding / plasma membrane-derived thylakoid membrane / chloride ion binding / plasma membrane-derived thylakoid membrane /  photoreceptor activity / photoreceptor activity /  光合成 / 光合成 /  lyase activity lyase activity類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア) | |||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2 Å クライオ電子顕微鏡法 / 解像度: 2 Å | |||||||||||||||
 データ登録者 データ登録者 | Sauer, P.V. / Sutter, M. / Cupellini, L. | |||||||||||||||
| 資金援助 | European Union,  米国, 米国,  チェコ, 4件 チェコ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2024 ジャーナル: Sci Adv / 年: 2024タイトル: Structural and quantum chemical basis for OCP-mediated quenching of phycobilisomes. 著者: Paul V Sauer / Lorenzo Cupellini / Markus Sutter / Mattia Bondanza / María Agustina Domínguez Martin / Henning Kirst / David Bína / Adrian Fujiet Koh / Abhay Kotecha / Basil J Greber / Eva ...著者: Paul V Sauer / Lorenzo Cupellini / Markus Sutter / Mattia Bondanza / María Agustina Domínguez Martin / Henning Kirst / David Bína / Adrian Fujiet Koh / Abhay Kotecha / Basil J Greber / Eva Nogales / Tomáš Polívka / Benedetta Mennucci / Cheryl A Kerfeld /      要旨: Cyanobacteria use large antenna complexes called phycobilisomes (PBSs) for light harvesting. However, intense light triggers non-photochemical quenching, where the orange carotenoid protein (OCP) ...Cyanobacteria use large antenna complexes called phycobilisomes (PBSs) for light harvesting. However, intense light triggers non-photochemical quenching, where the orange carotenoid protein (OCP) binds to PBS, dissipating excess energy as heat. The mechanism of efficiently transferring energy from phycocyanobilins in PBS to canthaxanthin in OCP remains insufficiently understood. Using cryo-electron microscopy, we unveiled the OCP-PBS complex structure at 1.6- to 2.1-angstrom resolution, showcasing its inherent flexibility. Using multiscale quantum chemistry, we disclosed the quenching mechanism. Identifying key protein residues, we clarified how canthaxanthin's transition dipole moment in its lowest-energy dark state becomes large enough for efficient energy transfer from phycocyanobilins. Our energy transfer model offers a detailed understanding of the atomic determinants of light harvesting regulation and antenna architecture in cyanobacteria. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8to2.cif.gz 8to2.cif.gz | 2.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8to2.ent.gz pdb8to2.ent.gz | 1.4 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8to2.json.gz 8to2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/to/8to2 https://data.pdbj.org/pub/pdb/validation_reports/to/8to2 ftp://data.pdbj.org/pub/pdb/validation_reports/to/8to2 ftp://data.pdbj.org/pub/pdb/validation_reports/to/8to2 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 3種, 3分子 ABp
| #1: タンパク質 | 分子量: 100412.703 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: Q55544 |
|---|---|
| #2: タンパク質 | 分子量: 34684.605 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: P74102 |
| #9: タンパク質 | 分子量: 12927.983 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: A0A6P1VGS4 |
-Phycobilisome ... , 2種, 3分子 CcP
| #3: タンパク質 | 分子量: 7817.174 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: Q02925 #6: タンパク質 | | 分子量: 28938.758 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: P73093 |
|---|
-Allophycocyanin ... , 4種, 23分子 DFHJLNfhjlEGIKMOegikmdo
| #4: タンパク質 | 分子量: 17429.807 Da / 分子数: 10 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: Q01951 #5: タンパク質 | 分子量: 17231.604 Da / 分子数: 11 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: Q01952 #7: タンパク質 | |  分子量: 17940.393 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: P72870 #8: タンパク質 | |  分子量: 18911.424 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Synechocystis sp. PCC 6803 (バクテリア) Synechocystis sp. PCC 6803 (バクテリア)参照: UniProt: P74551 |
|---|
-非ポリマー , 2種, 25分子 


| #10: 化合物 | ChemComp-CYC /  フィコシアノビリン フィコシアノビリン#11: 化合物 | ChemComp-45D / |  カンタキサンチン カンタキサンチン |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Complex of phycobilisome from Synechocystis PCC 6803 bound to OCP タイプ: COMPLEX / Entity ID: #1-#9 / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  |
| 緩衝液 | pH: 7.5 |
| 試料 | 濃度: 3.8 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 詳細: Manual blotting |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1200 nm / 最小 デフォーカス(公称値): 400 nm Bright-field microscopy / 最大 デフォーカス(公称値): 1200 nm / 最小 デフォーカス(公称値): 400 nm |
| 試料ホルダ | 凍結剤: NITROGEN |
| 撮影 | 電子線照射量: 40 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 画像処理 | 詳細: Streptavidin lattice was subtracted after movie alignment | ||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
3次元再構成 | 解像度: 2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 153576 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 19.17 Å2 | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj


