+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7yqk | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of gammaH2AXK15ub-H4K20me2 nucleosome bound to 53BP1 | ||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||
 キーワード キーワード |  NUCLEAR PROTEIN / nucleosoeme / NUCLEAR PROTEIN / nucleosoeme /  53BP1 / 53BP1 /  complex / H4K20me2 / H2AXK15ub / gammaH2AX complex / H4K20me2 / H2AXK15ub / gammaH2AX | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) ...Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication / DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) ...Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines / RMTs methylate histone arginines / DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / 遺伝的組換え / Pre-NOTCH Transcription and Translation /  nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin /  ヌクレオソーム / RUNX1 regulates transcription of genes involved in differentiation of HSCs / ヌクレオソーム / RUNX1 regulates transcription of genes involved in differentiation of HSCs /  遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression / 遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression /  nuclear body / nuclear body /  cadherin binding / protein heterodimerization activity / Amyloid fiber formation / DNA damage response / protein-containing complex / cadherin binding / protein heterodimerization activity / Amyloid fiber formation / DNA damage response / protein-containing complex /  DNA binding / extracellular exosome / extracellular region / DNA binding / extracellular exosome / extracellular region /  核質 / 核質 /  生体膜 / 生体膜 /  細胞核 細胞核類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | ||||||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.38 Å クライオ電子顕微鏡法 / 解像度: 3.38 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Ai, H.S. / GuoChao, C. / Qingyue, G. / Ze-Bin, T. / Zhiheng, D. / Xin, L. / Fan, Y. / Ziyu, X. / Jia-Bin, L. / Changlin, T. / Liu, L. | ||||||||||||||||||
| 資金援助 |  中国, 5件 中国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2022 ジャーナル: J Am Chem Soc / 年: 2022タイトル: Chemical Synthesis of Post-Translationally Modified H2AX Reveals Redundancy in Interplay between Histone Phosphorylation, Ubiquitination, and Methylation on the Binding of 53BP1 with Nucleosomes. 著者: Huasong Ai / Guo-Chao Chu / Qingyue Gong / Ze-Bin Tong / Zhiheng Deng / Xin Liu / Fan Yang / Ziyu Xu / Jia-Bin Li / Changlin Tian / Lei Liu /  要旨: The chemical synthesis of homogeneously modified histones is a powerful approach to quantitatively decipher how post-translational modifications (PTMs) modulate epigenetic events. Herein, we describe ...The chemical synthesis of homogeneously modified histones is a powerful approach to quantitatively decipher how post-translational modifications (PTMs) modulate epigenetic events. Herein, we describe the expedient syntheses of a selection of phosphorylated and ubiquitinated H2AX proteins in a strategy integrating expressed protein hydrazinolysis and auxiliary-mediated protein ligation. These modified H2AX proteins were then used to discover that although H2AXS139 phosphorylation can enhance the binding of the DNA damage repair factor 53BP1 to either an unmodified nucleosome or that bearing a single H2AXK15ub or H4K20me2 modification, it augments 53BP1's binding only weakly to nucleosomes bearing both H2AXK15ub and H4K20me2. To better understand why such a trivalent additive effect is lacking, we solved the cryo-EM structure (3.38 Å) of the complex of 53BP1 with the H2AXK15ub/S139ph_H4K20me2 nucleosome, which showed that H2AXS139 phosphorylation distorts the interaction interface between ubiquitin and 53BP1's UDR motif. Our study revealed that there is redundancy in the interplay of multiple histone PTMs, which may be useful for controlling the dynamic distribution of effector proteins onto nucleosomes bearing different histone variants and PTMs in a time-dependent fashion, through specific cellular biochemical events. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7yqk.cif.gz 7yqk.cif.gz | 313.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7yqk.ent.gz pdb7yqk.ent.gz | 242.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7yqk.json.gz 7yqk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yq/7yqk https://data.pdbj.org/pub/pdb/validation_reports/yq/7yqk ftp://data.pdbj.org/pub/pdb/validation_reports/yq/7yqk ftp://data.pdbj.org/pub/pdb/validation_reports/yq/7yqk | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | 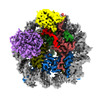 34028MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 6種, 10分子 AEBFCGDHON
| #1: タンパク質 |  ヒストンH3 / Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone ...Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone H3/i / Histone H3/j / Histone H3/k / Histone H3/l ヒストンH3 / Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone ...Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone H3/i / Histone H3/j / Histone H3/k / Histone H3/l分子量: 11762.835 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: H3C1, H3FA, HIST1H3A, H3C2, H3FL, HIST1H3B, H3C3, H3FC HIST1H3C, H3C4, H3FB, HIST1H3D, H3C6, H3FD, HIST1H3E, H3C7, H3FI, HIST1H3F, H3C8, H3FH, HIST1H3G, H3C10, H3FK, HIST1H3H, H3C11, H3FF, ...遺伝子: H3C1, H3FA, HIST1H3A, H3C2, H3FL, HIST1H3B, H3C3, H3FC HIST1H3C, H3C4, H3FB, HIST1H3D, H3C6, H3FD, HIST1H3E, H3C7, H3FI, HIST1H3F, H3C8, H3FH, HIST1H3G, H3C10, H3FK, HIST1H3H, H3C11, H3FF, HIST1H3I, H3C12, H3FJ, HIST1H3J 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P68431 Escherichia coli (大腸菌) / 参照: UniProt: P68431#2: タンパク質 |  ヒストンH4 ヒストンH4分子量: 9409.056 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: A0A672PXK1 Escherichia coli (大腸菌) / 参照: UniProt: A0A672PXK1#3: タンパク質 | 分子量: 12194.211 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) / 参照: UniProt: A0A6P5QZC6 #4: タンパク質 |  ヒストンH2B ヒストンH2B分子量: 10764.366 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: Q2M2T1 Escherichia coli (大腸菌) / 参照: UniProt: Q2M2T1#7: タンパク質 | | 分子量: 8519.778 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: UBB / 発現宿主: Homo sapiens (ヒト) / 遺伝子: UBB / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: J3QS39 Escherichia coli (大腸菌) / 参照: UniProt: J3QS39#9: タンパク質 | | 分子量: 13447.231 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: TP53BP1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TP53BP1 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: A6NNK5 Escherichia coli (大腸菌) / 参照: UniProt: A6NNK5 |
|---|
-DNA鎖 , 2種, 2分子 IJ
| #5: DNA鎖 | 分子量: 44520.383 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) |
|---|---|
| #6: DNA鎖 | 分子量: 44991.660 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) |
-タンパク質・ペプチド , 1種, 1分子 K
| #8: タンパク質・ペプチド | 分子量: 2075.418 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Complex of 53BP1(1484-1972) and gammaH2AXK15ub-H4K20me2 nucleosome タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE / 湿度: 100 % |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm / Cs Bright-field microscopy / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm / Cs : 2.7 mm : 2.7 mm |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.19.2_4158: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
3次元再構成 | 解像度: 3.38 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 90830 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj










































