+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7uos | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of WNK1 inhibitor complex | ||||||
 要素 要素 | Serine/threonine-protein kinase WNK1 | ||||||
 キーワード キーワード | TRANSFERASE/INHIBITOR / WNK1 /  inhibitor (酵素阻害剤) / trihalosulfone / inhibitor (酵素阻害剤) / trihalosulfone /  TRANSFERASE (転移酵素) / TRANSFERASE-INHIBITOR complex TRANSFERASE (転移酵素) / TRANSFERASE-INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of cell-cell adhesion mediated by integrin / lymphocyte migration into lymph node / chemokine (C-C motif) ligand 21 signaling pathway / positive regulation of termination of RNA polymerase II transcription / monoatomic cation homeostasis / negative regulation of pancreatic juice secretion / protein insertion into ER membrane by stop-transfer membrane-anchor sequence / positive regulation of mitotic cytokinesis / negative regulation of sodium ion transport / monoatomic ion homeostasis ...negative regulation of cell-cell adhesion mediated by integrin / lymphocyte migration into lymph node / chemokine (C-C motif) ligand 21 signaling pathway / positive regulation of termination of RNA polymerase II transcription / monoatomic cation homeostasis / negative regulation of pancreatic juice secretion / protein insertion into ER membrane by stop-transfer membrane-anchor sequence / positive regulation of mitotic cytokinesis / negative regulation of sodium ion transport / monoatomic ion homeostasis / positive regulation of potassium ion import across plasma membrane / regulation of mRNA export from nucleus / regulation of sodium ion transmembrane transport / intracellular chloride ion homeostasis / non-membrane-bounded organelle assembly / negative regulation of heterotypic cell-cell adhesion / positive regulation of T cell chemotaxis / positive regulation of systemic arterial blood pressure / potassium channel inhibitor activity / potassium ion homeostasis / cellular hyperosmotic response / cellular response to chemokine / protein serine/threonine kinase inhibitor activity / negative regulation of leukocyte cell-cell adhesion / regulation of monoatomic cation transmembrane transport / cell volume homeostasis / negative regulation of protein localization to plasma membrane / protein kinase activator activity / intracellular non-membrane-bounded organelle / sodium ion transmembrane transport / GABA-ergic synapse /  phosphatase binding / monoatomic ion transport / regulation of sodium ion transport / negative regulation of protein ubiquitination / cellular response to calcium ion / molecular condensate scaffold activity / negative regulation of autophagy / modulation of chemical synaptic transmission / phosphatase binding / monoatomic ion transport / regulation of sodium ion transport / negative regulation of protein ubiquitination / cellular response to calcium ion / molecular condensate scaffold activity / negative regulation of autophagy / modulation of chemical synaptic transmission /  紡錘体 / 紡錘体 /  血圧 / positive regulation of canonical Wnt signaling pathway / positive regulation of angiogenesis / 血圧 / positive regulation of canonical Wnt signaling pathway / positive regulation of angiogenesis /  heart development / T cell receptor signaling pathway / heart development / T cell receptor signaling pathway /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  protein kinase activity / intracellular signal transduction / protein kinase activity / intracellular signal transduction /  protein phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / protein phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity /  protein kinase binding / magnesium ion binding / protein kinase binding / magnesium ion binding /  シグナル伝達 / protein-containing complex / シグナル伝達 / protein-containing complex /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.9 Å 分子置換 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Akella, R. / Goldsmith, E.J. / Akella, R. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of WNK1 inhibitor complex 著者: Akella, R. / Goldsmith, E.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7uos.cif.gz 7uos.cif.gz | 74.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7uos.ent.gz pdb7uos.ent.gz | 52.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7uos.json.gz 7uos.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uo/7uos https://data.pdbj.org/pub/pdb/validation_reports/uo/7uos ftp://data.pdbj.org/pub/pdb/validation_reports/uo/7uos ftp://data.pdbj.org/pub/pdb/validation_reports/uo/7uos | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  5drbS S: 精密化の開始モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 33454.469 Da / 分子数: 1 / 変異: S382A / 由来タイプ: 組換発現 / 由来: (組換発現)   Rattus norvegicus (ドブネズミ) / 遺伝子: Wnk1, Hsn2, Prkwnk1 Rattus norvegicus (ドブネズミ) / 遺伝子: Wnk1, Hsn2, Prkwnk1発現宿主:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌)参照: UniProt: Q9JIH7,  non-specific serine/threonine protein kinase non-specific serine/threonine protein kinase | ||||
|---|---|---|---|---|---|
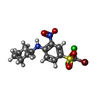
| #2: 化合物 | ChemComp-WIB / | ||||
| #3: 化合物 |  エチレングリコール エチレングリコール#4: 水 | ChemComp-HOH / |  水 水研究の焦点であるリガンドがあるか | Y | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.32 Å3/Da / 溶媒含有率: 47 % / 解説: plate |
|---|---|
結晶化 | 温度: 289 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.4 / 詳細: 20% PEG3350, 200mM Sodium Formate |
-データ収集
| 回折 | 平均測定温度: 163 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 0.97925 Å / ビームライン: 19-ID / 波長: 0.97925 Å |
| 検出器 | タイプ: APEX II CCD / 検出器: CCD / 日付: 2020年7月18日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.97925 Å / 相対比: 1 : 0.97925 Å / 相対比: 1 |
| 反射 | 解像度: 2.9→37.85 Å / Num. obs: 4894 / % possible obs: 86 % / 冗長度: 4.6 % / Biso Wilson estimate: 26.3 Å2 / CC1/2: 0.95 / Rpim(I) all: 0.06 / Rsym value: 0.12 / Net I/σ(I): 10.4 |
| 反射 シェル | 解像度: 2.9→2.9 Å / Num. unique obs: 4894 / CC1/2: 0.95 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 5drb 解像度: 2.9→37.85 Å / Cor.coef. Fo:Fc: 0.876 / Cor.coef. Fo:Fc free: 0.768 / SU B: 24.545 / SU ML: 0.455 / 交差検証法: THROUGHOUT / ESU R Free: 0.637 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.9 Å / 減衰半径: 0.9 Å / VDWプローブ半径: 1 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 49.901 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.9→37.85 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj