+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7ty8 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human Anion Exchanger 1 bound to Niflumic Acid | ||||||||||||
 要素 要素 | Band 3 anion transport protein | ||||||||||||
 キーワード キーワード |  TRANSPORT PROTEIN (運搬体タンパク質) / TRANSPORT PROTEIN (運搬体タンパク質) /  Transmembrane (膜貫通型タンパク質) Transmembrane (膜貫通型タンパク質) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / Bicarbonate transporters / intracellular monoatomic ion homeostasis /  ankyrin-1 complex / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / solute:inorganic anion antiporter activity ...pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / Bicarbonate transporters / intracellular monoatomic ion homeostasis / ankyrin-1 complex / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / solute:inorganic anion antiporter activity ...pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / Bicarbonate transporters / intracellular monoatomic ion homeostasis /  ankyrin-1 complex / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / solute:inorganic anion antiporter activity / bicarbonate transmembrane transporter activity / bicarbonate transport / monoatomic anion transport / chloride transmembrane transporter activity / chloride transport / erythrocyte development / negative regulation of glycolytic process through fructose-6-phosphate / ankyrin-1 complex / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / solute:inorganic anion antiporter activity / bicarbonate transmembrane transporter activity / bicarbonate transport / monoatomic anion transport / chloride transmembrane transporter activity / chloride transport / erythrocyte development / negative regulation of glycolytic process through fructose-6-phosphate /  ankyrin binding / ankyrin binding /  hemoglobin binding / cortical cytoskeleton / protein-membrane adaptor activity / chloride transmembrane transport / protein localization to plasma membrane / hemoglobin binding / cortical cytoskeleton / protein-membrane adaptor activity / chloride transmembrane transport / protein localization to plasma membrane /  regulation of intracellular pH / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / transmembrane transport / cytoplasmic side of plasma membrane / Z disc / regulation of intracellular pH / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / transmembrane transport / cytoplasmic side of plasma membrane / Z disc /  凝固・線溶系 / basolateral plasma membrane / blood microparticle / protein homodimerization activity / extracellular exosome / 凝固・線溶系 / basolateral plasma membrane / blood microparticle / protein homodimerization activity / extracellular exosome /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.18 Å クライオ電子顕微鏡法 / 解像度: 3.18 Å | ||||||||||||
 データ登録者 データ登録者 | Capper, M.J. / Mathiharan, Y.K. / Yang, S. / Stone, A.C. / Wacker, D. | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Substrate binding and inhibition of the anion exchanger 1 transporter. 著者: Michael J Capper / Shifan Yang / Alexander C Stone / Sezen Vatansever / Gregory Zilberg / Yamuna Kalyani Mathiharan / Raul Habib / Keino Hutchinson / Yihan Zhao / Avner Schlessinger / Mihaly ...著者: Michael J Capper / Shifan Yang / Alexander C Stone / Sezen Vatansever / Gregory Zilberg / Yamuna Kalyani Mathiharan / Raul Habib / Keino Hutchinson / Yihan Zhao / Avner Schlessinger / Mihaly Mezei / Roman Osman / Bin Zhang / Daniel Wacker /  要旨: Anion exchanger 1 (AE1), a member of the solute carrier (SLC) family, is the primary bicarbonate transporter in erythrocytes, regulating pH levels and CO transport between lungs and tissues. Previous ...Anion exchanger 1 (AE1), a member of the solute carrier (SLC) family, is the primary bicarbonate transporter in erythrocytes, regulating pH levels and CO transport between lungs and tissues. Previous studies characterized its role in erythrocyte structure and provided insight into transport regulation. However, key questions remain regarding substrate binding and transport, mechanisms of drug inhibition and modulation by membrane components. Here we present seven cryo-EM structures in apo, bicarbonate-bound and inhibitor-bound states. These, combined with uptake and computational studies, reveal important molecular features of substrate recognition and transport, and illuminate sterol binding sites, to elucidate distinct inhibitory mechanisms of research chemicals and prescription drugs. We further probe the substrate binding site via structure-based ligand screening, identifying an AE1 inhibitor. Together, our findings provide insight into mechanisms of solute carrier transport and inhibition. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7ty8.cif.gz 7ty8.cif.gz | 241 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7ty8.ent.gz pdb7ty8.ent.gz | 180.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7ty8.json.gz 7ty8.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ty/7ty8 https://data.pdbj.org/pub/pdb/validation_reports/ty/7ty8 ftp://data.pdbj.org/pub/pdb/validation_reports/ty/7ty8 ftp://data.pdbj.org/pub/pdb/validation_reports/ty/7ty8 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  26169MC  7ty4C  7ty6C  7ty7C  7tyaC  8t6uC  8t6vC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Ens-ID: 1 / Beg auth comp-ID: ASP / Beg label comp-ID: ASP / End auth comp-ID: ALA / End label comp-ID: ALA / Auth seq-ID: 370 - 891 / Label seq-ID: 370 - 891
NCSアンサンブル: (詳細: Global NCS restraints between domains: 1 2) NCS oper:
|
- 要素
要素
-タンパク質 / 糖 , 2種, 4分子 AB
| #1: タンパク質 |  / Anion exchange protein 1 / AE 1 / Anion exchanger 1 / Solute carrier family 4 member 1 / Anion exchange protein 1 / AE 1 / Anion exchanger 1 / Solute carrier family 4 member 1分子量: 101883.859 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SLC4A1, AE1, DI, EPB3 / プラスミド: pFB / 細胞株 (発現宿主): Sf9 Homo sapiens (ヒト) / 遺伝子: SLC4A1, AE1, DI, EPB3 / プラスミド: pFB / 細胞株 (発現宿主): Sf9発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P02730 #2: 多糖 |  オリゴ糖 / 分子量: 424.401 Da / 分子数: 2 / 由来タイプ: 組換発現 オリゴ糖 / 分子量: 424.401 Da / 分子数: 2 / 由来タイプ: 組換発現 |
|---|
-非ポリマー , 4種, 41分子 

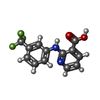




| #3: 化合物 | ChemComp-CLR /  コレステロール コレステロール#4: 化合物 |  ホスファチジルコリン ホスファチジルコリン#5: 化合物 |  Niflumic acid Niflumic acid#6: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Dimeric anion exchanger 1 (SLC4A1) / タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.203 MDa / 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)株: Sf9 / プラスミド  : pFB : pFB | ||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||
| 試料 | 濃度: 5 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: The sample was monodisperse following gel filtration and immediately concentrated for grid preparation | ||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||
急速凍結 | 装置: LEICA EM GP / 凍結剤: ETHANE / 湿度: 95 % / 凍結前の試料温度: 285 K 詳細: Blot force 3 for 3-5 seconds was used and subsequent grids were screened for ice thickness prior to data collection |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 64000 X / 最大 デフォーカス(公称値): 1800 nm / 最小 デフォーカス(公称値): 500 nm / Cs Bright-field microscopy / 倍率(公称値): 64000 X / 最大 デフォーカス(公称値): 1800 nm / 最小 デフォーカス(公称値): 500 nm / Cs : 2.7 mm : 2.7 mm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 59.99 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 5274 |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0267 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2977492 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C2 (2回回転対称 : C2 (2回回転対称 ) ) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.18 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 79981 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 空間: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 4YZF Accession code: 4YZF / Source name: PDB / タイプ: experimental model | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.18→133.424 Å / Cor.coef. Fo:Fc: 0.874 / WRfactor Rwork: 0.414 / Average fsc overall: 0.7467 / Average fsc work: 0.7467 / ESU R: 0.623 / 詳細: Hydrogens have been added in their riding positions
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: BABINET MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 94.249 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj









