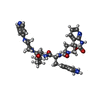
| 登録情報 | データベース: PDB / ID: 6zho
|
|---|
| タイトル | Crystal structure of a CGRP receptor ectodomain heterodimer with bound high affinity inhibitor |
|---|
 要素 要素 | Maltose/maltodextrin-binding periplasmic protein,Receptor activity-modifying protein 1,Calcitonin gene-related peptide type 1 receptor |
|---|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  GPCR (Gタンパク質共役受容体) / GPCR (Gタンパク質共役受容体) /  CGRP (カルシトニン遺伝子関連ペプチド) / CLR / RAMP1 / ECD / CGRP (カルシトニン遺伝子関連ペプチド) / CLR / RAMP1 / ECD /  COMPLEX COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
 calcitonin gene-related peptide binding / calcitonin gene-related peptide binding /  CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway / CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway /  adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus / adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus /  adrenomedullin receptor activity / adrenomedullin receptor activity /  adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity ... adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity ... calcitonin gene-related peptide binding / calcitonin gene-related peptide binding /  CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway / CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway /  adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus / adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus /  adrenomedullin receptor activity / adrenomedullin receptor activity /  adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity / adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity /  calcitonin receptor activity / vascular associated smooth muscle cell proliferation / calcitonin receptor activity / vascular associated smooth muscle cell proliferation /  calcitonin gene-related peptide receptor activity / amylin receptor signaling pathway / Calcitonin-like ligand receptors / regulation of G protein-coupled receptor signaling pathway / detection of maltose stimulus / calcitonin gene-related peptide receptor activity / amylin receptor signaling pathway / Calcitonin-like ligand receptors / regulation of G protein-coupled receptor signaling pathway / detection of maltose stimulus /  maltose binding / maltose transport complex / maltose transport / maltodextrin transmembrane transport / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / carbohydrate transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / carbohydrate transport / maltose binding / maltose transport complex / maltose transport / maltodextrin transmembrane transport / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / carbohydrate transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / carbohydrate transport /  coreceptor activity / cellular response to hormone stimulus / positive regulation of vascular associated smooth muscle cell proliferation / ATP-binding cassette (ABC) transporter complex / cell chemotaxis / G protein-coupled receptor activity / protein localization to plasma membrane / coreceptor activity / cellular response to hormone stimulus / positive regulation of vascular associated smooth muscle cell proliferation / ATP-binding cassette (ABC) transporter complex / cell chemotaxis / G protein-coupled receptor activity / protein localization to plasma membrane /  intracellular protein transport / adenylate cyclase-activating G protein-coupled receptor signaling pathway / intracellular protein transport / adenylate cyclase-activating G protein-coupled receptor signaling pathway /  receptor internalization / calcium ion transport / receptor internalization / calcium ion transport /  protein transport / outer membrane-bounded periplasmic space / protein transport / outer membrane-bounded periplasmic space /  heart development / G alpha (s) signalling events / heart development / G alpha (s) signalling events /  血管新生 / 血管新生 /  リソソーム / リソソーム /  ペリプラズム / cell surface receptor signaling pathway / ペリプラズム / cell surface receptor signaling pathway /  receptor complex / receptor complex /  エンドソーム / G protein-coupled receptor signaling pathway / DNA damage response / エンドソーム / G protein-coupled receptor signaling pathway / DNA damage response /  細胞膜 / 細胞膜 /  小胞体 / 小胞体 /  生体膜 / 生体膜 /  細胞膜 / 細胞膜 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 GPCR, family 2, calcitonin gene-related peptide, type 1 receptor / Receptor activity modifying protein / RAMP domain superfamily / Receptor activity modifying family / GPCR, family 2, calcitonin gene-related peptide, type 1 receptor / Receptor activity modifying protein / RAMP domain superfamily / Receptor activity modifying family /  GPCR, family 2, calcitonin receptor family / G-protein coupled receptors family 2 signature 1. / GPCR, family 2, calcitonin receptor family / G-protein coupled receptors family 2 signature 1. /  Hormone receptor domain / Hormone receptor domain /  GPCR, family 2, extracellular hormone receptor domain / G-protein coupled receptors family 2 profile 1. / Domain present in hormone receptors ... GPCR, family 2, extracellular hormone receptor domain / G-protein coupled receptors family 2 profile 1. / Domain present in hormone receptors ... GPCR, family 2, calcitonin gene-related peptide, type 1 receptor / Receptor activity modifying protein / RAMP domain superfamily / Receptor activity modifying family / GPCR, family 2, calcitonin gene-related peptide, type 1 receptor / Receptor activity modifying protein / RAMP domain superfamily / Receptor activity modifying family /  GPCR, family 2, calcitonin receptor family / G-protein coupled receptors family 2 signature 1. / GPCR, family 2, calcitonin receptor family / G-protein coupled receptors family 2 signature 1. /  Hormone receptor domain / Hormone receptor domain /  GPCR, family 2, extracellular hormone receptor domain / G-protein coupled receptors family 2 profile 1. / Domain present in hormone receptors / GPCR family 2, extracellular hormone receptor domain superfamily / G-protein coupled receptors family 2 signature 2. / GPCR, family 2, extracellular hormone receptor domain / G-protein coupled receptors family 2 profile 1. / Domain present in hormone receptors / GPCR family 2, extracellular hormone receptor domain superfamily / G-protein coupled receptors family 2 signature 2. /  GPCR, family 2, secretin-like, conserved site / GPCR, family 2, secretin-like, conserved site /  GPCR, family 2, secretin-like / 7 transmembrane receptor (Secretin family) / GPCR, family 2, secretin-like / 7 transmembrane receptor (Secretin family) /  GPCR, family 2-like / G-protein coupled receptors family 2 profile 2. / Maltose/Cyclodextrin ABC transporter, substrate-binding protein / Solute-binding family 1, conserved site / Bacterial extracellular solute-binding proteins, family 1 signature. / Bacterial extracellular solute-binding protein / Bacterial extracellular solute-binding protein類似検索 - ドメイン・相同性 GPCR, family 2-like / G-protein coupled receptors family 2 profile 2. / Maltose/Cyclodextrin ABC transporter, substrate-binding protein / Solute-binding family 1, conserved site / Bacterial extracellular solute-binding proteins, family 1 signature. / Bacterial extracellular solute-binding protein / Bacterial extracellular solute-binding protein類似検索 - ドメイン・相同性alpha-maltose / Chem-QLQ / Receptor activity-modifying protein 1 / Maltose/maltodextrin-binding periplasmic protein / Calcitonin gene-related peptide type 1 receptor類似検索 - 構成要素 |
|---|
| 生物種 |    Escherichia coli (大腸菌) Escherichia coli (大腸菌)
  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.6 Å 分子置換 / 解像度: 1.6 Å |
|---|
 データ登録者 データ登録者 | Southall, S.M. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2020 ジャーナル: J.Med.Chem. / 年: 2020
タイトル: Structure-Based Drug Discovery ofN-((R)-3-(7-Methyl-1H-indazol-5-yl)-1-oxo-1-(((S)-1-oxo-3-(piperidin-4-yl)-1-(4-(pyridin-4-yl)piperazin-1-yl)propan-2-yl)amino)propan-2-yl)-2'-oxo-1',2'- ...タイトル: Structure-Based Drug Discovery ofN-((R)-3-(7-Methyl-1H-indazol-5-yl)-1-oxo-1-(((S)-1-oxo-3-(piperidin-4-yl)-1-(4-(pyridin-4-yl)piperazin-1-yl)propan-2-yl)amino)propan-2-yl)-2'-oxo-1',2'-dihydrospiro[piperidine-4,4'-pyrido[2,3-d][1,3]oxazine]-1-carboxamide (HTL22562): A Calcitonin Gene-Related Peptide Receptor Antagonist for Acute Treatment of Migraine.
著者: Bucknell, S.J. / Ator, M.A. / Brown, A.J.H. / Brown, J. / Cansfield, A.D. / Cansfield, J.E. / Christopher, J.A. / Congreve, M. / Cseke, G. / Deflorian, F. / Jones, C.R. / Mason, J.S. / ...著者: Bucknell, S.J. / Ator, M.A. / Brown, A.J.H. / Brown, J. / Cansfield, A.D. / Cansfield, J.E. / Christopher, J.A. / Congreve, M. / Cseke, G. / Deflorian, F. / Jones, C.R. / Mason, J.S. / O'Brien, M.A. / Ott, G.R. / Pickworth, M. / Southall, S.M. |
|---|
| 履歴 | | 登録 | 2020年6月23日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2020年7月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年7月22日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.title / _citation_author.identifier_ORCID / _citation_author.name |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / atom_site_anisotrop ...atom_site / atom_site_anisotrop / chem_comp / entity / entity_name_com / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_molecule_features / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _atom_site_anisotrop.U[1][1] / _atom_site_anisotrop.U[1][2] / _atom_site_anisotrop.U[1][3] / _atom_site_anisotrop.U[2][2] / _atom_site_anisotrop.U[2][3] / _atom_site_anisotrop.U[3][3] / _atom_site_anisotrop.pdbx_auth_asym_id / _atom_site_anisotrop.pdbx_auth_atom_id / _atom_site_anisotrop.pdbx_auth_comp_id / _atom_site_anisotrop.pdbx_auth_seq_id / _atom_site_anisotrop.pdbx_label_asym_id / _atom_site_anisotrop.pdbx_label_atom_id / _atom_site_anisotrop.pdbx_label_comp_id / _atom_site_anisotrop.type_symbol / _chem_comp.name / _entity.formula_weight / _entity.pdbx_description / _entity.pdbx_number_of_molecules / _entity.src_method / _entity.type / _pdbx_struct_assembly_gen.asym_id_list / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2020年8月5日 | Group: Database references / Structure summary / カテゴリ: chem_comp / citation / citation_author
Item: _chem_comp.pdbx_synonyms / _citation.journal_volume ..._chem_comp.pdbx_synonyms / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID |
|---|
| 改定 3.0 | 2021年8月18日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Database references / Derived calculations / Polymer sequence / Source and taxonomy / Structure summary
カテゴリ: atom_site / atom_site_anisotrop ...atom_site / atom_site_anisotrop / database_2 / entity / entity_poly / entity_poly_seq / entity_src_gen / pdbx_nonpoly_scheme / pdbx_poly_seq_scheme / pdbx_struct_sheet_hbond / pdbx_unobs_or_zero_occ_residues / pdbx_validate_close_contact / pdbx_validate_polymer_linkage / pdbx_validate_symm_contact / struct_conf / struct_conn / struct_ref / struct_ref_seq / struct_ref_seq_dif / struct_sheet_range
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.label_seq_id / _atom_site_anisotrop.U[1][1] / _atom_site_anisotrop.U[1][2] / _atom_site_anisotrop.U[1][3] / _atom_site_anisotrop.U[2][2] / _atom_site_anisotrop.U[2][3] / _atom_site_anisotrop.U[3][3] / _atom_site_anisotrop.pdbx_label_seq_id / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _entity.formula_weight / _entity_poly.pdbx_seq_one_letter_code / _entity_poly.pdbx_seq_one_letter_code_can / _entity_src_gen.gene_src_strain / _entity_src_gen.pdbx_beg_seq_num / _entity_src_gen.pdbx_end_seq_num / _pdbx_nonpoly_scheme.auth_seq_num / _pdbx_struct_sheet_hbond.range_1_label_seq_id / _pdbx_struct_sheet_hbond.range_2_label_seq_id / _pdbx_validate_close_contact.auth_seq_id_1 / _pdbx_validate_close_contact.auth_seq_id_2 / _pdbx_validate_symm_contact.auth_seq_id_1 / _pdbx_validate_symm_contact.auth_seq_id_2 / _struct_conf.beg_label_seq_id / _struct_conf.end_label_seq_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_label_seq_id / _struct_ref.pdbx_align_begin / _struct_ref.pdbx_seq_one_letter_code / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end / _struct_ref_seq.pdbx_auth_seq_align_beg / _struct_ref_seq.pdbx_auth_seq_align_end / _struct_ref_seq.seq_align_beg / _struct_ref_seq.seq_align_end / _struct_sheet_range.beg_label_seq_id / _struct_sheet_range.end_label_seq_id |
|---|
| 改定 3.1 | 2021年8月25日 | Group: Advisory / Database references / Structure summary
カテゴリ: pdbx_poly_seq_scheme / pdbx_unobs_or_zero_occ_residues / struct_ref_seq
Item: _pdbx_poly_seq_scheme.pdb_seq_num / _pdbx_unobs_or_zero_occ_residues.auth_seq_id / _struct_ref_seq.pdbx_auth_seq_align_end |
|---|
| 改定 3.2 | 2024年1月24日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード MEMBRANE PROTEIN (膜タンパク質) /
MEMBRANE PROTEIN (膜タンパク質) /  GPCR (Gタンパク質共役受容体) /
GPCR (Gタンパク質共役受容体) /  CGRP (カルシトニン遺伝子関連ペプチド) / CLR / RAMP1 / ECD /
CGRP (カルシトニン遺伝子関連ペプチド) / CLR / RAMP1 / ECD /  COMPLEX
COMPLEX 機能・相同性情報
機能・相同性情報 calcitonin gene-related peptide binding /
calcitonin gene-related peptide binding /  CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway /
CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway /  adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus /
adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus /  adrenomedullin receptor activity /
adrenomedullin receptor activity /  adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity ...
adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity ... calcitonin gene-related peptide binding /
calcitonin gene-related peptide binding /  CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway /
CGRP receptor complex / calcitonin gene-related peptide receptor signaling pathway /  adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus /
adrenomedullin binding / positive regulation of protein glycosylation / cellular response to sucrose stimulus /  adrenomedullin receptor activity /
adrenomedullin receptor activity /  adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity /
adrenomedullin receptor complex / adrenomedullin receptor signaling pathway / amylin receptor activity /  calcitonin receptor activity / vascular associated smooth muscle cell proliferation /
calcitonin receptor activity / vascular associated smooth muscle cell proliferation /  calcitonin gene-related peptide receptor activity / amylin receptor signaling pathway / Calcitonin-like ligand receptors / regulation of G protein-coupled receptor signaling pathway / detection of maltose stimulus /
calcitonin gene-related peptide receptor activity / amylin receptor signaling pathway / Calcitonin-like ligand receptors / regulation of G protein-coupled receptor signaling pathway / detection of maltose stimulus /  maltose binding / maltose transport complex / maltose transport / maltodextrin transmembrane transport / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / carbohydrate transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / carbohydrate transport /
maltose binding / maltose transport complex / maltose transport / maltodextrin transmembrane transport / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / carbohydrate transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / carbohydrate transport /  coreceptor activity / cellular response to hormone stimulus / positive regulation of vascular associated smooth muscle cell proliferation / ATP-binding cassette (ABC) transporter complex / cell chemotaxis / G protein-coupled receptor activity / protein localization to plasma membrane /
coreceptor activity / cellular response to hormone stimulus / positive regulation of vascular associated smooth muscle cell proliferation / ATP-binding cassette (ABC) transporter complex / cell chemotaxis / G protein-coupled receptor activity / protein localization to plasma membrane /  intracellular protein transport / adenylate cyclase-activating G protein-coupled receptor signaling pathway /
intracellular protein transport / adenylate cyclase-activating G protein-coupled receptor signaling pathway /  receptor internalization / calcium ion transport /
receptor internalization / calcium ion transport /  protein transport / outer membrane-bounded periplasmic space /
protein transport / outer membrane-bounded periplasmic space /  heart development / G alpha (s) signalling events /
heart development / G alpha (s) signalling events /  血管新生 /
血管新生 /  リソソーム /
リソソーム /  ペリプラズム / cell surface receptor signaling pathway /
ペリプラズム / cell surface receptor signaling pathway /  receptor complex /
receptor complex /  エンドソーム / G protein-coupled receptor signaling pathway / DNA damage response /
エンドソーム / G protein-coupled receptor signaling pathway / DNA damage response /  細胞膜 /
細胞膜 /  小胞体 /
小胞体 /  生体膜 /
生体膜 /  細胞膜 /
細胞膜 /  細胞質
細胞質
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2020
ジャーナル: J.Med.Chem. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6zho.cif.gz
6zho.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6zho.ent.gz
pdb6zho.ent.gz PDB形式
PDB形式 6zho.json.gz
6zho.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/zh/6zho
https://data.pdbj.org/pub/pdb/validation_reports/zh/6zho ftp://data.pdbj.org/pub/pdb/validation_reports/zh/6zho
ftp://data.pdbj.org/pub/pdb/validation_reports/zh/6zho リンク
リンク 集合体
集合体
 要素
要素
 Escherichia coli (strain K12) (大腸菌), (組換発現)
Escherichia coli (strain K12) (大腸菌), (組換発現) 
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト) / 参照: UniProt: P0AEX9, UniProt: O60894, UniProt: Q16602
Homo sapiens (ヒト) / 参照: UniProt: P0AEX9, UniProt: O60894, UniProt: Q16602 ポリエチレングリコール
ポリエチレングリコール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I24 / 波長: 0.96861 Å
/ ビームライン: I24 / 波長: 0.96861 Å : 0.96861 Å / 相対比: 1
: 0.96861 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj