+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6yx5 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of DrrA from Legionella pneumophilia in complex with human Rab8a | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | CELL INVASION / vesicular trafficking virulence factor | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / protein guanylylation / neurotransmitter receptor transport to postsynaptic membrane / Golgi vesicle fusion to target membrane / AMPylase activity / vesicle-mediated transport in synapse / protein adenylyltransferase /  regulation of protein transport / protein adenylylation / VxPx cargo-targeting to cilium ...: / protein guanylylation / neurotransmitter receptor transport to postsynaptic membrane / Golgi vesicle fusion to target membrane / AMPylase activity / vesicle-mediated transport in synapse / protein adenylyltransferase / regulation of protein transport / protein adenylylation / VxPx cargo-targeting to cilium ...: / protein guanylylation / neurotransmitter receptor transport to postsynaptic membrane / Golgi vesicle fusion to target membrane / AMPylase activity / vesicle-mediated transport in synapse / protein adenylyltransferase /  regulation of protein transport / protein adenylylation / VxPx cargo-targeting to cilium / neurotransmitter receptor transport, endosome to postsynaptic membrane / RAB geranylgeranylation / regulation of protein transport / protein adenylylation / VxPx cargo-targeting to cilium / neurotransmitter receptor transport, endosome to postsynaptic membrane / RAB geranylgeranylation /  myosin V binding / vesicle docking involved in exocytosis / myosin V binding / vesicle docking involved in exocytosis /  regulation of exocytosis / trans-Golgi network transport vesicle / RAB GEFs exchange GTP for GDP on RABs / protein localization to cilium / non-motile cilium / host cell cytoplasmic vesicle / endocytic recycling / phosphatidylinositol-4-phosphate binding / TBC/RABGAPs / ciliary membrane / ciliary base / regulation of exocytosis / trans-Golgi network transport vesicle / RAB GEFs exchange GTP for GDP on RABs / protein localization to cilium / non-motile cilium / host cell cytoplasmic vesicle / endocytic recycling / phosphatidylinositol-4-phosphate binding / TBC/RABGAPs / ciliary membrane / ciliary base /  regulation of GTPase activity / Golgi organization / regulation of GTPase activity / Golgi organization /  protein secretion / protein secretion /  cilium assembly / phagocytic vesicle / Anchoring of the basal body to the plasma membrane / cilium assembly / phagocytic vesicle / Anchoring of the basal body to the plasma membrane /  protein tyrosine kinase binding / protein tyrosine kinase binding /  中心小体 / 中心小体 /  軸索誘導 / guanyl-nucleotide exchange factor activity / 軸索誘導 / guanyl-nucleotide exchange factor activity /  低分子量GTPアーゼ / trans-Golgi network membrane / ciliary basal body / 低分子量GTPアーゼ / trans-Golgi network membrane / ciliary basal body /  regulation of autophagy / Translocation of SLC2A4 (GLUT4) to the plasma membrane / host cell cytoplasmic vesicle membrane / protein localization to plasma membrane / regulation of long-term neuronal synaptic plasticity / regulation of autophagy / Translocation of SLC2A4 (GLUT4) to the plasma membrane / host cell cytoplasmic vesicle membrane / protein localization to plasma membrane / regulation of long-term neuronal synaptic plasticity /  繊毛 / 繊毛 /  small GTPase binding / small GTPase binding /  オートファジー / cellular response to insulin stimulus / recycling endosome membrane / phagocytic vesicle membrane / GDP binding / オートファジー / cellular response to insulin stimulus / recycling endosome membrane / phagocytic vesicle membrane / GDP binding /  Regulation of PLK1 Activity at G2/M Transition / Regulation of PLK1 Activity at G2/M Transition /  シナプス小胞 / protein guanylyltransferase activity / midbody / シナプス小胞 / protein guanylyltransferase activity / midbody /  樹状突起スパイン / 樹状突起スパイン /  postsynaptic density / endosome membrane / postsynaptic density / endosome membrane /  エンドソーム / エンドソーム /  ゴルジ体 / ゴルジ体 /  GTPase activity / GTPase activity /  中心体 / neuronal cell body / glutamatergic synapse / GTP binding / extracellular exosome / extracellular region / 中心体 / neuronal cell body / glutamatergic synapse / GTP binding / extracellular exosome / extracellular region /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)  Legionella pneumophila (レジオネラ・ニューモフィラ) Legionella pneumophila (レジオネラ・ニューモフィラ) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.14 Å 分子置換 / 解像度: 2.14 Å | |||||||||
 データ登録者 データ登録者 | Schneider, S. / Du, J. / von Wrisberg, M.K. / Lang, K. / Itzen, A. | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Rab1-AMPylation by Legionella DrrA is allosterically activated by Rab1. 著者: Du, J. / Wrisberg, M.V. / Gulen, B. / Stahl, M. / Pett, C. / Hedberg, C. / Lang, K. / Schneider, S. / Itzen, A. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6yx5.cif.gz 6yx5.cif.gz | 227.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6yx5.ent.gz pdb6yx5.ent.gz | 180.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6yx5.json.gz 6yx5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yx/6yx5 https://data.pdbj.org/pub/pdb/validation_reports/yx/6yx5 ftp://data.pdbj.org/pub/pdb/validation_reports/yx/6yx5 ftp://data.pdbj.org/pub/pdb/validation_reports/yx/6yx5 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 20042.062 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: recombinant / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: Rab8a / 発現宿主: Homo sapiens (ヒト) / 遺伝子: Rab8a / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: A0A1U7REJ8, UniProt: P61006*PLUS Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: A0A1U7REJ8, UniProt: P61006*PLUS |
|---|---|
| #2: タンパク質 | 分子量: 38837.301 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: residue number 1 disordered L197C 由来: (組換発現)   Legionella pneumophila (レジオネラ・ニューモフィラ) Legionella pneumophila (レジオネラ・ニューモフィラ)発現宿主:   Escherichia coli BL21 (大腸菌) / 参照: UniProt: Q29ST3 Escherichia coli BL21 (大腸菌) / 参照: UniProt: Q29ST3 |
-糖 , 1種, 3分子 
| #5: 糖 |  グルコース グルコース |
|---|
-非ポリマー , 5種, 121分子 

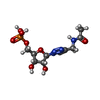






| #3: 化合物 | ChemComp-MG / | ||
|---|---|---|---|
| #4: 化合物 | ChemComp-GNP /  5'-Guanylyl imidodiphosphate 5'-Guanylyl imidodiphosphate | ||
| #6: 化合物 | ChemComp-LJN / [( | ||
| #7: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩#8: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.8 Å3/Da / 溶媒含有率: 45 % / 解説: hexagonal |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 9 / 詳細: 0.1M bicine, pH 9, 2.4M ammonium sulphate |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: MASSIF-1 / 波長: 0.966 Å / ビームライン: MASSIF-1 / 波長: 0.966 Å |
| 検出器 | タイプ: DECTRIS PILATUS 2M / 検出器: PIXEL / 日付: 2018年9月7日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.966 Å / 相対比: 1 : 0.966 Å / 相対比: 1 |
| 反射 | 解像度: 2.14→48.06 Å / Num. obs: 49091 / % possible obs: 99.7 % / 冗長度: 20 % / Biso Wilson estimate: 43.52 Å2 / CC1/2: 0.99 / CC star: 1 / Rmerge(I) obs: 0.2 / Rpim(I) all: 0.045 / Rrim(I) all: 0.206 / Net I/σ(I): 15.57 |
| 反射 シェル | 解像度: 2.14→2.22 Å / 冗長度: 18.3 % / Rmerge(I) obs: 2.43 / Mean I/σ(I) obs: 0.93 / Num. unique obs: 4745 / CC1/2: 0.43 / CC star: 0.775 / Rpim(I) all: 0.57 / Rrim(I) all: 2.5 / % possible all: 96.97 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 3NKU, 4LHV 解像度: 2.14→48.06 Å / Cor.coef. Fo:Fc: 0.956 / Cor.coef. Fo:Fc free: 0.941 / SU B: 11.681 / SU ML: 0.143 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.174 / ESU R Free: 0.159 詳細: U VALUES : WITH TLS ADDED HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.7 Å / 減衰半径: 0.7 Å / VDWプローブ半径: 1 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 113.26 Å2 / Biso mean: 50.306 Å2 / Biso min: 25.38 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.14→48.06 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.144→2.2 Å / Rfactor Rfree error: 0
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj
















