+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6avu | ||||||
|---|---|---|---|---|---|---|---|
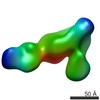
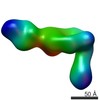
| タイトル | Human alpha-V beta-3 Integrin (open conformation) in complex with the therapeutic antibody LM609 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  SIGNALING PROTEIN / alpha-V beta-3 integrin / LM609 / SIGNALING PROTEIN / alpha-V beta-3 integrin / LM609 /  vitaxin / abegrin vitaxin / abegrin | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報integrin alphav-beta6 complex / integrin alphav-beta8 complex / transforming growth factor beta production / negative regulation of entry of bacterium into host cell / integrin alphav-beta5 complex / : /  opsonin binding / integrin alphav-beta1 complex / Cross-presentation of particulate exogenous antigens (phagosomes) / opsonin binding / integrin alphav-beta1 complex / Cross-presentation of particulate exogenous antigens (phagosomes) /  extracellular matrix protein binding ...integrin alphav-beta6 complex / integrin alphav-beta8 complex / transforming growth factor beta production / negative regulation of entry of bacterium into host cell / integrin alphav-beta5 complex / : / extracellular matrix protein binding ...integrin alphav-beta6 complex / integrin alphav-beta8 complex / transforming growth factor beta production / negative regulation of entry of bacterium into host cell / integrin alphav-beta5 complex / : /  opsonin binding / integrin alphav-beta1 complex / Cross-presentation of particulate exogenous antigens (phagosomes) / opsonin binding / integrin alphav-beta1 complex / Cross-presentation of particulate exogenous antigens (phagosomes) /  extracellular matrix protein binding / tube development / regulation of serotonin uptake / positive regulation of adenylate cyclase-inhibiting opioid receptor signaling pathway / alpha9-beta1 integrin-ADAM8 complex / regulation of trophoblast cell migration / regulation of postsynaptic neurotransmitter receptor diffusion trapping / alphav-beta3 integrin-vitronectin complex / maintenance of postsynaptic specialization structure / Laminin interactions / positive regulation of glomerular mesangial cell proliferation / regulation of extracellular matrix organization / platelet alpha granule membrane / integrin alphav-beta3 complex / negative regulation of lipoprotein metabolic process / entry into host cell by a symbiont-containing vacuole / alphav-beta3 integrin-PKCalpha complex / extracellular matrix protein binding / tube development / regulation of serotonin uptake / positive regulation of adenylate cyclase-inhibiting opioid receptor signaling pathway / alpha9-beta1 integrin-ADAM8 complex / regulation of trophoblast cell migration / regulation of postsynaptic neurotransmitter receptor diffusion trapping / alphav-beta3 integrin-vitronectin complex / maintenance of postsynaptic specialization structure / Laminin interactions / positive regulation of glomerular mesangial cell proliferation / regulation of extracellular matrix organization / platelet alpha granule membrane / integrin alphav-beta3 complex / negative regulation of lipoprotein metabolic process / entry into host cell by a symbiont-containing vacuole / alphav-beta3 integrin-PKCalpha complex /  fibrinogen binding / glycinergic synapse / alphav-beta3 integrin-HMGB1 complex / fibrinogen binding / glycinergic synapse / alphav-beta3 integrin-HMGB1 complex /  vascular endothelial growth factor receptor 2 binding / vascular endothelial growth factor receptor 2 binding /  blood coagulation, fibrin clot formation / negative regulation of lipid transport / negative regulation of low-density lipoprotein receptor activity / Elastic fibre formation / blood coagulation, fibrin clot formation / negative regulation of lipid transport / negative regulation of low-density lipoprotein receptor activity / Elastic fibre formation /  regulation of phagocytosis / cell-substrate junction assembly / regulation of release of sequestered calcium ion into cytosol / mesodermal cell differentiation / alphav-beta3 integrin-IGF-1-IGF1R complex / regulation of phagocytosis / cell-substrate junction assembly / regulation of release of sequestered calcium ion into cytosol / mesodermal cell differentiation / alphav-beta3 integrin-IGF-1-IGF1R complex /  transforming growth factor beta binding / angiogenesis involved in wound healing / transforming growth factor beta binding / angiogenesis involved in wound healing /  platelet-derived growth factor receptor binding / filopodium membrane / positive regulation of small GTPase mediated signal transduction / platelet-derived growth factor receptor binding / filopodium membrane / positive regulation of small GTPase mediated signal transduction /  extracellular matrix binding / positive regulation of fibroblast migration / positive regulation of vascular endothelial growth factor receptor signaling pathway / regulation of postsynaptic neurotransmitter receptor internalization / apolipoprotein A-I-mediated signaling pathway / extracellular matrix binding / positive regulation of fibroblast migration / positive regulation of vascular endothelial growth factor receptor signaling pathway / regulation of postsynaptic neurotransmitter receptor internalization / apolipoprotein A-I-mediated signaling pathway /  regulation of bone resorption / regulation of bone resorption /  wound healing, spreading of epidermal cells / apoptotic cell clearance / heterotypic cell-cell adhesion / wound healing, spreading of epidermal cells / apoptotic cell clearance / heterotypic cell-cell adhesion /  integrin complex / positive regulation of cell adhesion mediated by integrin / Molecules associated with elastic fibres / cellular response to insulin-like growth factor stimulus / positive regulation of intracellular signal transduction / positive regulation of cell-matrix adhesion / cell adhesion mediated by integrin / smooth muscle cell migration / microvillus membrane / Syndecan interactions / negative chemotaxis / p130Cas linkage to MAPK signaling for integrins / cellular response to platelet-derived growth factor stimulus / integrin complex / positive regulation of cell adhesion mediated by integrin / Molecules associated with elastic fibres / cellular response to insulin-like growth factor stimulus / positive regulation of intracellular signal transduction / positive regulation of cell-matrix adhesion / cell adhesion mediated by integrin / smooth muscle cell migration / microvillus membrane / Syndecan interactions / negative chemotaxis / p130Cas linkage to MAPK signaling for integrins / cellular response to platelet-derived growth factor stimulus /  protein disulfide isomerase activity / cell-substrate adhesion / positive regulation of smooth muscle cell migration / endodermal cell differentiation / activation of protein kinase activity / positive regulation of osteoblast proliferation / TGF-beta receptor signaling activates SMADs / PECAM1 interactions / lamellipodium membrane / GRB2:SOS provides linkage to MAPK signaling for Integrins / negative regulation of macrophage derived foam cell differentiation / negative regulation of lipid storage / platelet-derived growth factor receptor signaling pathway / protein disulfide isomerase activity / cell-substrate adhesion / positive regulation of smooth muscle cell migration / endodermal cell differentiation / activation of protein kinase activity / positive regulation of osteoblast proliferation / TGF-beta receptor signaling activates SMADs / PECAM1 interactions / lamellipodium membrane / GRB2:SOS provides linkage to MAPK signaling for Integrins / negative regulation of macrophage derived foam cell differentiation / negative regulation of lipid storage / platelet-derived growth factor receptor signaling pathway /  fibronectin binding / positive regulation of cell adhesion / ECM proteoglycans / fibronectin binding / positive regulation of cell adhesion / ECM proteoglycans /  voltage-gated calcium channel activity / positive regulation of T cell migration / positive regulation of bone resorption / voltage-gated calcium channel activity / positive regulation of T cell migration / positive regulation of bone resorption /  脈管形成 / Integrin cell surface interactions / specific granule membrane / 脈管形成 / Integrin cell surface interactions / specific granule membrane /  coreceptor activity / phagocytic vesicle / negative regulation of endothelial cell apoptotic process / positive regulation of substrate adhesion-dependent cell spreading / coreceptor activity / phagocytic vesicle / negative regulation of endothelial cell apoptotic process / positive regulation of substrate adhesion-dependent cell spreading /  着床 / extrinsic apoptotic signaling pathway in absence of ligand / ERK1 and ERK2 cascade / positive regulation of endothelial cell proliferation / 着床 / extrinsic apoptotic signaling pathway in absence of ligand / ERK1 and ERK2 cascade / positive regulation of endothelial cell proliferation /  cell adhesion molecule binding / Integrin signaling / substrate adhesion-dependent cell spreading / cell-matrix adhesion cell adhesion molecule binding / Integrin signaling / substrate adhesion-dependent cell spreading / cell-matrix adhesion類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)  Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 35 Å ネガティブ染色法 / 解像度: 35 Å | ||||||
 データ登録者 データ登録者 | Borst, A.J. / James, Z.N. / Zagotta, W.N. / Ginsberg, M. / Rey, F.A. / DiMaio, F. / Backovic, M. / Veesler, D. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2017 ジャーナル: Structure / 年: 2017タイトル: The Therapeutic Antibody LM609 Selectively Inhibits Ligand Binding to Human αβ Integrin via Steric Hindrance. 著者: Andrew J Borst / Zachary M James / William N Zagotta / Mark Ginsberg / Felix A Rey / Frank DiMaio / Marija Backovic / David Veesler /   要旨: The LM609 antibody specifically recognizes αβ integrin and inhibits angiogenesis, bone resorption, and viral infections in an arginine-glycine-aspartate-independent manner. LM609 entered phase II ...The LM609 antibody specifically recognizes αβ integrin and inhibits angiogenesis, bone resorption, and viral infections in an arginine-glycine-aspartate-independent manner. LM609 entered phase II clinical trials for the treatment of several cancers and was also used for αβ-targeted radioimmunotherapy. To elucidate the mechanisms of recognition and inhibition of αβ integrin, we solved the structure of the LM609 antigen-binding fragment by X-ray crystallography and determined its binding affinity for αβ. Using single-particle electron microscopy, we show that LM609 binds at the interface between the β-propeller domain of the α chain and the βI domain of the β chain, near the RGD-binding site, of all observed integrin conformational states. Integrating these data with fluorescence size-exclusion chromatography, we demonstrate that LM609 sterically hinders access of large ligands to the RGD-binding pocket, without obstructing it. This work provides a structural framework to expedite future efforts utilizing LM609 as a diagnostic or therapeutic tool. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6avu.cif.gz 6avu.cif.gz | 280.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6avu.ent.gz pdb6avu.ent.gz | 189.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6avu.json.gz 6avu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/av/6avu https://data.pdbj.org/pub/pdb/validation_reports/av/6avu ftp://data.pdbj.org/pub/pdb/validation_reports/av/6avu ftp://data.pdbj.org/pub/pdb/validation_reports/av/6avu | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  Integrin alpha V / Vitronectin receptor / Vitronectin receptor subunit alpha Integrin alpha V / Vitronectin receptor / Vitronectin receptor subunit alpha分子量: 105894.188 Da / 分子数: 1 / 断片: UNP residues 31-987 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: ITGAV, MSK8, VNRA, VTNR Homo sapiens (ヒト) / 遺伝子: ITGAV, MSK8, VNRA, VTNR発現宿主:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ)参照: UniProt: P06756 |
|---|---|
| #2: タンパク質 |  Integrin beta 3 / Platelet membrane glycoprotein IIIa / GPIIIa Integrin beta 3 / Platelet membrane glycoprotein IIIa / GPIIIa分子量: 76523.125 Da / 分子数: 1 / 断片: UNP residues 27-718 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: ITGB3, GP3A Homo sapiens (ヒト) / 遺伝子: ITGB3, GP3A発現宿主:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ)参照: UniProt: P05106 |
| #3: 抗体 | 分子量: 27223.100 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ)発現宿主:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| #4: 抗体 | 分子量: 23628.977 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ)発現宿主:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Quaternary complex of human alpha-V beta-3 integrin with the Fab LM609 タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.23 MDa / 実験値: NO |
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 緩衝液 | pH: 8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : YES / 凍結 : YES / 凍結 : NO : NO |
| 染色 | タイプ: NEGATIVE / 染色剤: Uranyl formate |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: C-flat 2/0.5 |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: FEI TECNAI 12 |
|---|---|
| 電子銃 | 電子線源 : LAB6 / 加速電圧: 120 kV / 照射モード: FLOOD BEAM : LAB6 / 加速電圧: 120 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | 電子線照射量: 30 e/Å2 フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) |
- 解析
解析
| EMソフトウェア |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: NONE | |||||||||
3次元再構成 | 解像度: 35 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 650 / 対称性のタイプ: POINT | |||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj













