| 登録情報 | データベース: PDB / ID: 5w8z
|
|---|
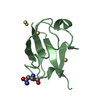
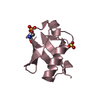
| タイトル | Solution Structure of XPH2, a Hybrid Sequence of Xfaso 1 and Pfl 6, Two Cro Proteins With Different Folds |
|---|
 要素 要素 | XPH2 |
|---|
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / TRANSCRIPTION (転写 (生物学)) /  helix-turn-helix (ヘリックスターンヘリックス) / structural evolution / conformational switch / helix-turn-helix (ヘリックスターンヘリックス) / structural evolution / conformational switch /  metamorphic protein (変成岩) / metamorphic protein (変成岩) /  disulfide bond (ジスルフィド) / disulfide bond (ジスルフィド) /  transcription factor (転写因子) transcription factor (転写因子) |
|---|
| 機能・相同性 | lambda repressor-like DNA-binding domains / 434 Repressor (Amino-terminal Domain) / Orthogonal Bundle / Mainly Alpha 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Pseudomonas fluorescens PF5 (蛍光菌) Pseudomonas fluorescens PF5 (蛍光菌) |
|---|
| 手法 |  溶液NMR / CYANA 溶液NMR / CYANA |
|---|
 データ登録者 データ登録者 | Kumirov, V.K. / Dykstra, E.M. / Cordes, M.H. |
|---|
| 資金援助 |  米国, 2件 米国, 2件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM066806 |  米国 米国 | | National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM104040 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Protein Sci. / 年: 2018 ジャーナル: Protein Sci. / 年: 2018
タイトル: Multistep mutational transformation of a protein fold through structural intermediates.
著者: Kumirov, V.K. / Dykstra, E.M. / Hall, B.M. / Anderson, W.J. / Szyszka, T.N. / Cordes, M.H.J. |
|---|
| 履歴 | | 登録 | 2017年6月22日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2018年7月11日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年8月8日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.name |
|---|
| 改定 1.2 | 2018年11月7日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.3 | 2020年1月1日 | Group: Author supporting evidence / Data collection / カテゴリ: pdbx_audit_support / pdbx_nmr_software
Item: _pdbx_audit_support.funding_organization / _pdbx_nmr_software.name |
|---|
| 改定 1.4 | 2023年6月14日 | Group: Database references / Other / カテゴリ: database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSCRIPTION (転写 (生物学)) /
TRANSCRIPTION (転写 (生物学)) /  helix-turn-helix (ヘリックスターンヘリックス) / structural evolution / conformational switch /
helix-turn-helix (ヘリックスターンヘリックス) / structural evolution / conformational switch /  metamorphic protein (変成岩) /
metamorphic protein (変成岩) /  disulfide bond (ジスルフィド) /
disulfide bond (ジスルフィド) /  transcription factor (転写因子)
transcription factor (転写因子) 機能・相同性情報
機能・相同性情報
 Pseudomonas fluorescens PF5 (蛍光菌)
Pseudomonas fluorescens PF5 (蛍光菌) 溶液NMR / CYANA
溶液NMR / CYANA  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Protein Sci. / 年: 2018
ジャーナル: Protein Sci. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5w8z.cif.gz
5w8z.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5w8z.ent.gz
pdb5w8z.ent.gz PDB形式
PDB形式 5w8z.json.gz
5w8z.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/w8/5w8z
https://data.pdbj.org/pub/pdb/validation_reports/w8/5w8z ftp://data.pdbj.org/pub/pdb/validation_reports/w8/5w8z
ftp://data.pdbj.org/pub/pdb/validation_reports/w8/5w8z
 リンク
リンク 集合体
集合体
 要素
要素
 Pseudomonas fluorescens PF5 (蛍光菌)
Pseudomonas fluorescens PF5 (蛍光菌)
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) 溶液NMR
溶液NMR 試料調製
試料調製 : INOVA / 磁場強度: 600 MHz
: INOVA / 磁場強度: 600 MHz 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj