| 登録情報 | データベース: PDB / ID: 5tbn
|
|---|
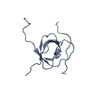
| タイトル | Solution NMR structure of PHF20 PHD domain in complex with a histone H3K4me2 peptide |
|---|
 要素 要素 | - Histone H3.1
 ヒストンH3 ヒストンH3 - PHD finger protein 20

|
|---|
 キーワード キーワード | TRANSCRIPTION / STRUCTURAL PROTEIN /  PHD finger / Methylated lysine / TRANSCRIPTION - STRUCTURAL PROTEIN complex PHD finger / Methylated lysine / TRANSCRIPTION - STRUCTURAL PROTEIN complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
NSL complex / : / Regulation of TP53 Activity through Association with Co-factors / Formation of WDR5-containing histone-modifying complexes /  MLL1 complex / MLL1 complex /  histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening ...NSL complex / : / Regulation of TP53 Activity through Association with Co-factors / Formation of WDR5-containing histone-modifying complexes / histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening ...NSL complex / : / Regulation of TP53 Activity through Association with Co-factors / Formation of WDR5-containing histone-modifying complexes /  MLL1 complex / MLL1 complex /  histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication / histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Stabilization of p53 / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines / RMTs methylate histone arginines / DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Stabilization of p53 / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation / 遺伝的組換え / Pre-NOTCH Transcription and Translation /  nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin /  ヌクレオソーム / Regulation of TP53 Degradation / RUNX1 regulates transcription of genes involved in differentiation of HSCs / ヌクレオソーム / Regulation of TP53 Degradation / RUNX1 regulates transcription of genes involved in differentiation of HSCs /  遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence / 遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence /  核膜 / Estrogen-dependent gene expression / 核膜 / Estrogen-dependent gene expression /  cadherin binding / protein heterodimerization activity / Amyloid fiber formation / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / protein-containing complex / cadherin binding / protein heterodimerization activity / Amyloid fiber formation / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / protein-containing complex /  DNA binding / extracellular exosome / extracellular region / DNA binding / extracellular exosome / extracellular region /  核質 / 核質 /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質類似検索 - 分子機能 細胞質基質類似検索 - 分子機能  PHD finger protein 20, AT hook / PHD finger protein 20, AT hook /  PHD finger protein 20, AT-hook / PHD finger protein 20, AT-hook /  DNA repair protein Crb2, Tudor domain / DNA repair protein Crb2, Tudor domain /  DNA repair protein Crb2 Tudor domain / DNA repair protein Crb2 Tudor domain /  PHD finger protein 20-like / PHD finger protein 20-like /  PhD finger domain / PhD finger domain /  Tudor domain / Tudor domain /  Tudor domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 ... Tudor domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 ... PHD finger protein 20, AT hook / PHD finger protein 20, AT hook /  PHD finger protein 20, AT-hook / PHD finger protein 20, AT-hook /  DNA repair protein Crb2, Tudor domain / DNA repair protein Crb2, Tudor domain /  DNA repair protein Crb2 Tudor domain / DNA repair protein Crb2 Tudor domain /  PHD finger protein 20-like / PHD finger protein 20-like /  PhD finger domain / PhD finger domain /  Tudor domain / Tudor domain /  Tudor domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / Zinc finger C2H2 type domain profile. / Tudor domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / Zinc finger C2H2 type domain profile. /  Zinc finger, PHD-type, conserved site / Zinc finger C2H2 type domain signature. / Zinc finger PHD-type signature. / Zinc finger C2H2-type / Histone H3 signature 1. / Zinc finger, PHD-type, conserved site / Zinc finger C2H2 type domain signature. / Zinc finger PHD-type signature. / Zinc finger C2H2-type / Histone H3 signature 1. /  Zinc finger, PHD-type / PHD zinc finger / Histone H3 signature 2. / Zinc finger, PHD-type / PHD zinc finger / Histone H3 signature 2. /  ヒストンH3 / Histone H3/CENP-A / Histone H2A/H2B/H3 / Core histone H2A/H2B/H3/H4 / ヒストンH3 / Histone H3/CENP-A / Histone H2A/H2B/H3 / Core histone H2A/H2B/H3/H4 /  Zinc finger, FYVE/PHD-type / Histone-fold / Zinc finger, FYVE/PHD-type / Histone-fold /  Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / 溶液NMR /  simulated annealing simulated annealing |
|---|
 データ登録者 データ登録者 | Cui, G. / Botuyan, M.V. / Mer, G. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Cancer Institute (NIH/NCI) | CA132878 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Cell Rep / 年: 2016 ジャーナル: Cell Rep / 年: 2016
タイトル: PHF20 Readers Link Methylation of Histone H3K4 and p53 with H4K16 Acetylation.
著者: Klein, B.J. / Wang, X. / Cui, G. / Yuan, C. / Botuyan, M.V. / Lin, K. / Lu, Y. / Wang, X. / Zhao, Y. / Bruns, C.J. / Mer, G. / Shi, X. / Kutateladze, T.G. |
|---|
| 履歴 | | 登録 | 2016年9月12日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年10月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2016年11月2日 | Group: Database references |
|---|
| 改定 1.2 | 2017年9月20日 | Group: Author supporting evidence / Structure summary / カテゴリ: entity / pdbx_audit_support
Item: _entity.pdbx_number_of_molecules / _pdbx_audit_support.funding_organization |
|---|
| 改定 1.3 | 2019年12月4日 | Group: Author supporting evidence / Data collection
カテゴリ: pdbx_audit_support / pdbx_nmr_software / pdbx_nmr_spectrometer
Item: _pdbx_audit_support.funding_organization / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード PHD finger / Methylated lysine / TRANSCRIPTION - STRUCTURAL PROTEIN complex
PHD finger / Methylated lysine / TRANSCRIPTION - STRUCTURAL PROTEIN complex 機能・相同性情報
機能・相同性情報 MLL1 complex /
MLL1 complex /  histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening ...NSL complex / : / Regulation of TP53 Activity through Association with Co-factors / Formation of WDR5-containing histone-modifying complexes /
histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening ...NSL complex / : / Regulation of TP53 Activity through Association with Co-factors / Formation of WDR5-containing histone-modifying complexes /  MLL1 complex /
MLL1 complex /  histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /
histone acetyltransferase complex / Chromatin modifying enzymes / epigenetic regulation of gene expression / telomere organization / RNA Polymerase I Promoter Opening / Interleukin-7 signaling / Assembly of the ORC complex at the origin of replication /  DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Stabilization of p53 / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines / RMTs methylate histone arginines /
DNAメチル化 / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / PRC2 methylates histones and DNA / Defective pyroptosis / HDACs deacetylate histones / RNA Polymerase I Promoter Escape / Stabilization of p53 / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / HDMs demethylate histones / PKMTs methylate histone lysines / RMTs methylate histone arginines /  遺伝的組換え / Pre-NOTCH Transcription and Translation /
遺伝的組換え / Pre-NOTCH Transcription and Translation /  nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin /
nucleosome assembly / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / Transcriptional regulation of granulopoiesis / structural constituent of chromatin /  ヌクレオソーム / Regulation of TP53 Degradation / RUNX1 regulates transcription of genes involved in differentiation of HSCs /
ヌクレオソーム / Regulation of TP53 Degradation / RUNX1 regulates transcription of genes involved in differentiation of HSCs /  遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence /
遺伝子発現 / chromatin organization / Factors involved in megakaryocyte development and platelet production / HATs acetylate histones / Senescence-Associated Secretory Phenotype (SASP) / Oxidative Stress Induced Senescence /  核膜 / Estrogen-dependent gene expression /
核膜 / Estrogen-dependent gene expression /  cadherin binding / protein heterodimerization activity / Amyloid fiber formation / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / protein-containing complex /
cadherin binding / protein heterodimerization activity / Amyloid fiber formation / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / protein-containing complex /  DNA binding / extracellular exosome / extracellular region /
DNA binding / extracellular exosome / extracellular region /  核質 /
核質 /  生体膜 /
生体膜 /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞質基質
細胞質基質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR /
溶液NMR /  simulated annealing
simulated annealing  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Cell Rep / 年: 2016
ジャーナル: Cell Rep / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5tbn.cif.gz
5tbn.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5tbn.ent.gz
pdb5tbn.ent.gz PDB形式
PDB形式 5tbn.json.gz
5tbn.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/tb/5tbn
https://data.pdbj.org/pub/pdb/validation_reports/tb/5tbn ftp://data.pdbj.org/pub/pdb/validation_reports/tb/5tbn
ftp://data.pdbj.org/pub/pdb/validation_reports/tb/5tbn
 リンク
リンク 集合体
集合体
 要素
要素 / Glioma-expressed antigen 2 / Hepatocellular carcinoma-associated antigen 58 / Novel zinc finger ...Glioma-expressed antigen 2 / Hepatocellular carcinoma-associated antigen 58 / Novel zinc finger protein / Transcription factor TZP
/ Glioma-expressed antigen 2 / Hepatocellular carcinoma-associated antigen 58 / Novel zinc finger ...Glioma-expressed antigen 2 / Hepatocellular carcinoma-associated antigen 58 / Novel zinc finger protein / Transcription factor TZP
 Homo sapiens (ヒト) / 遺伝子: PHF20, C20orf104, GLEA2, HCA58, NZF, TZP / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PHF20, C20orf104, GLEA2, HCA58, NZF, TZP / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q9BVI0
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q9BVI0 ヒストンH3 / Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone ...Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone H3/i / Histone H3/j / Histone H3/k / Histone H3/l
ヒストンH3 / Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone ...Histone H3/a / Histone H3/b / Histone H3/c / Histone H3/d / Histone H3/f / Histone H3/h / Histone H3/i / Histone H3/j / Histone H3/k / Histone H3/l
 Homo sapiens (ヒト) / 参照: UniProt: P68431
Homo sapiens (ヒト) / 参照: UniProt: P68431 溶液NMR
溶液NMR 試料調製
試料調製 : AVANCE III / 磁場強度: 700 MHz / 詳細: Cryoprobe
: AVANCE III / 磁場強度: 700 MHz / 詳細: Cryoprobe 解析
解析 simulated annealing / ソフトェア番号: 9
simulated annealing / ソフトェア番号: 9  ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj
















