+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5qaq | ||||||
|---|---|---|---|---|---|---|---|
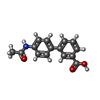
| タイトル | OXA-48 IN COMPLEX WITH COMPOUND 21b | ||||||
 要素 要素 | Beta-lactamase Β-ラクタマーゼ Β-ラクタマーゼ | ||||||
 キーワード キーワード |  HYDROLASE (加水分解酵素) / Oxacillinase / Inhibitor / HYDROLASE (加水分解酵素) / Oxacillinase / Inhibitor /  Complex / OXA / Complex / OXA /  Antibiotic resistance (抗微生物薬耐性) / Antibiotic resistance (抗微生物薬耐性) /  beta-lactamase (Β-ラクタマーゼ) / fragment beta-lactamase (Β-ラクタマーゼ) / fragment | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 penicillin binding / antibiotic catabolic process / penicillin binding / antibiotic catabolic process /  beta-lactamase activity / beta-lactamase activity /  Β-ラクタマーゼ / response to antibiotic Β-ラクタマーゼ / response to antibiotic類似検索 - 分子機能 | ||||||
| 生物種 |   Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.4 Å 分子置換 / 解像度: 2.4 Å | ||||||
 データ登録者 データ登録者 | Lund, B.A. / Leiros, H.K.S. | ||||||
 引用 引用 |  ジャーナル: Eur J Med Chem / 年: 2018 ジャーナル: Eur J Med Chem / 年: 2018タイトル: A focused fragment library targeting the antibiotic resistance enzyme - Oxacillinase-48: Synthesis, structural evaluation and inhibitor design. 著者: Akhter, S. / Lund, B.A. / Ismael, A. / Langer, M. / Isaksson, J. / Christopeit, T. / Leiros, H.S. / Bayer, A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5qaq.cif.gz 5qaq.cif.gz | 580.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5qaq.ent.gz pdb5qaq.ent.gz | 491.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5qaq.json.gz 5qaq.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qa/5qaq https://data.pdbj.org/pub/pdb/validation_reports/qa/5qaq ftp://data.pdbj.org/pub/pdb/validation_reports/qa/5qaq ftp://data.pdbj.org/pub/pdb/validation_reports/qa/5qaq | HTTPS FTP |
|---|
-グループ登録
| ID | G_1002027 (37エントリ) |
|---|---|
| タイトル | A focused fragment library targeting the antibiotic resistance enzyme - oxacillinase-48: synthesis, structural evaluation and inhibitor design |
| タイプ | undefined |
| 解説 | A focused fragment library targeting the antibiotic resistance enzyme - oxacillinase-48: synthesis, structural evaluation and inhibitor design |
-関連構造データ
| 関連構造データ |  5dtkS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Ens-ID: 1 / Beg auth comp-ID: GLU / Beg label comp-ID: GLU / End auth comp-ID: PRO / End label comp-ID: PRO / Auth seq-ID: 24 - 265 / Label seq-ID: 2 - 243
|
- 要素
要素
| #1: タンパク質 |  Β-ラクタマーゼ Β-ラクタマーゼ分子量: 28226.951 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)   Klebsiella pneumoniae (肺炎桿菌) / 遺伝子: bla OXA-48, blaOXA-48, KPE71T_00045 / 発現宿主: Klebsiella pneumoniae (肺炎桿菌) / 遺伝子: bla OXA-48, blaOXA-48, KPE71T_00045 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: Q6XEC0, Escherichia coli (大腸菌) / 参照: UniProt: Q6XEC0,  Β-ラクタマーゼ Β-ラクタマーゼ#2: 化合物 |  塩化物 塩化物#3: 化合物 | ChemComp-EDO /  エチレングリコール エチレングリコール#4: 化合物 | #5: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.68 Å3/Da / 溶媒含有率: 54.06 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 0.1 M HEPES pH 7.5, 8-11% PEG 8000 and 4-8% 1-butanol |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID29 / 波長: 0.97623 Å / ビームライン: ID29 / 波長: 0.97623 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2016年5月12日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.97623 Å / 相対比: 1 : 0.97623 Å / 相対比: 1 |
| 反射 | 解像度: 2.4→43.46 Å / % possible obs: 99.68 % / 冗長度: 7.3 % / Rmerge(I) obs: 0.09331 / Rrim(I) all: 0.1 / Net I/σ(I): 15.2 |
| 反射 シェル | 解像度: 2.4→2.49 Å / % possible obs: 99.54 % / 冗長度: 7.2 % / Rmerge(I) obs: 0.7104 / Num. unique obs: 34137 / Rrim(I) all: 0.77 / Net I/σ(I) obs: 2.56 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY: 5dtk 解像度: 2.4→43.46 Å / 交差検証法: THROUGHOUT
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.4→43.46 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 17
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj