+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5pz7 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | PanDDA analysis group deposition -- Crystal Structure of SP100 after initial refinement with no ligand modelled (structure 103) | ||||||
 要素 要素 | Nuclear autoantigen Sp-100 | ||||||
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / PanDDA / SGC - Diamond I04-1 fragment screening / TRANSCRIPTION (転写 (生物学)) / PanDDA / SGC - Diamond I04-1 fragment screening /  bromodomain (ブロモドメイン) / bromodomain (ブロモドメイン) /  epigenetics (エピジェネティクス) / XChemExplorer epigenetics (エピジェネティクス) / XChemExplorer | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of Fas signaling pathway / maintenance of protein location /  chromo shadow domain binding / negative regulation of viral transcription / response to type I interferon / negative regulation of protein export from nucleus / negative regulation of endothelial cell migration / regulation of extrinsic apoptotic signaling pathway via death domain receptors / type I interferon-mediated signaling pathway / response to type II interferon ...regulation of Fas signaling pathway / maintenance of protein location / chromo shadow domain binding / negative regulation of viral transcription / response to type I interferon / negative regulation of protein export from nucleus / negative regulation of endothelial cell migration / regulation of extrinsic apoptotic signaling pathway via death domain receptors / type I interferon-mediated signaling pathway / response to type II interferon ...regulation of Fas signaling pathway / maintenance of protein location /  chromo shadow domain binding / negative regulation of viral transcription / response to type I interferon / negative regulation of protein export from nucleus / negative regulation of endothelial cell migration / regulation of extrinsic apoptotic signaling pathway via death domain receptors / type I interferon-mediated signaling pathway / response to type II interferon / negative regulation of DNA binding / DNA damage response, signal transduction by p53 class mediator / retinoic acid receptor signaling pathway / chromo shadow domain binding / negative regulation of viral transcription / response to type I interferon / negative regulation of protein export from nucleus / negative regulation of endothelial cell migration / regulation of extrinsic apoptotic signaling pathway via death domain receptors / type I interferon-mediated signaling pathway / response to type II interferon / negative regulation of DNA binding / DNA damage response, signal transduction by p53 class mediator / retinoic acid receptor signaling pathway /  regulation of angiogenesis / SUMOylation of DNA damage response and repair proteins / type II interferon-mediated signaling pathway / response to retinoic acid / regulation of angiogenesis / SUMOylation of DNA damage response and repair proteins / type II interferon-mediated signaling pathway / response to retinoic acid /  telomere maintenance / nuclear periphery / response to cytokine / negative regulation of DNA-binding transcription factor activity / PML body / telomere maintenance / nuclear periphery / response to cytokine / negative regulation of DNA-binding transcription factor activity / PML body /  kinase binding / positive regulation of DNA-binding transcription factor activity / Interferon gamma signaling / RNA polymerase II-specific DNA-binding transcription factor binding / kinase binding / positive regulation of DNA-binding transcription factor activity / Interferon gamma signaling / RNA polymerase II-specific DNA-binding transcription factor binding /  chromosome, telomeric region / chromosome, telomeric region /  protein dimerization activity / protein dimerization activity /  nuclear body / DNA-binding transcription factor activity, RNA polymerase II-specific / protein domain specific binding / negative regulation of DNA-templated transcription / nuclear body / DNA-binding transcription factor activity, RNA polymerase II-specific / protein domain specific binding / negative regulation of DNA-templated transcription /  核小体 / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / 核小体 / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / positive regulation of transcription by RNA polymerase II /  DNA binding / DNA binding /  核質 / identical protein binding / 核質 / identical protein binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  フーリエ合成 / フーリエ合成 /  分子置換 / 解像度: 1.54 Å 分子置換 / 解像度: 1.54 Å | ||||||
 データ登録者 データ登録者 | Pearce, N.M. / Krojer, T. / Talon, R. / Bradley, A.R. / Fairhead, M. / Sethi, R. / Wright, N. / MacLean, E. / Collins, P. / Brandao-Neto, J. ...Pearce, N.M. / Krojer, T. / Talon, R. / Bradley, A.R. / Fairhead, M. / Sethi, R. / Wright, N. / MacLean, E. / Collins, P. / Brandao-Neto, J. / Douangamath, A. / Renjie, Z. / Dias, A. / Ng, J. / Brennan, P.E. / Cox, O. / Bountra, C. / Arrowsmith, C.H. / Edwards, A. / von Delft, F. | ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: A multi-crystal method for extracting obscured crystallographic states from conventionally uninterpretable electron density. 著者: Pearce, N.M. / Krojer, T. / Bradley, A.R. / Collins, P. / Nowak, R.P. / Talon, R. / Marsden, B.D. / Kelm, S. / Shi, J. / Deane, C.M. / von Delft, F. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
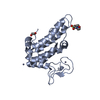
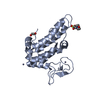
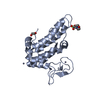
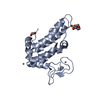
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5pz7.cif.gz 5pz7.cif.gz | 104.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5pz7.ent.gz pdb5pz7.ent.gz | 79.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5pz7.json.gz 5pz7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pz/5pz7 https://data.pdbj.org/pub/pdb/validation_reports/pz/5pz7 ftp://data.pdbj.org/pub/pdb/validation_reports/pz/5pz7 ftp://data.pdbj.org/pub/pdb/validation_reports/pz/5pz7 | HTTPS FTP |
|---|
-グループ登録
| ID | G_1002025 (114エントリ) |
|---|---|
| タイトル | PanDDA analysis group deposition of models of ground state datasets |
| タイプ | ground state |
| 解説 | human SP100 screened against the Maybridge Library by X-ray Crystallography at the XChem facility of Diamond Light Source beamline I04-1. Check out the PanDDA event maps at https://zenodo.org/record/290201/files/0_index.html |
-関連構造データ
| 関連構造データ |  4ptbS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 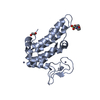
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 21498.564 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SP100 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SP100 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P23497 Escherichia coli (大腸菌) / 参照: UniProt: P23497#2: 化合物 | ChemComp-ZN / #3: 化合物 | ChemComp-MES / |  MES (緩衝剤) MES (緩衝剤)#4: 化合物 |  エチレングリコール エチレングリコール#5: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.76 Å3/Da / 溶媒含有率: 55.47 % / Mosaicity: 0 ° |
|---|---|
結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 / 詳細: 0.1M MES pH 6.1 -- 20% PEG20K |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I04-1 / 波長: 0.92 Å / ビームライン: I04-1 / 波長: 0.92 Å | |||||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS PILATUS 2M / 検出器: PIXEL / 日付: 2015年4月2日 | |||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.92 Å / 相対比: 1 : 0.92 Å / 相対比: 1 | |||||||||||||||||||||||||||
| 反射 | 解像度: 1.54→31.47 Å / Num. obs: 65960 / % possible obs: 94.8 % / 冗長度: 3.5 % / CC1/2: 0.999 / Rmerge(I) obs: 0.047 / Rpim(I) all: 0.029 / Rrim(I) all: 0.055 / Net I/σ(I): 13.6 / Num. measured all: 231816 / Scaling rejects: 0 | |||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / 冗長度: 3.5 %
|
-位相決定
位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  フーリエ合成 フーリエ合成開始モデル: 4PTB 解像度: 1.54→31.47 Å / Cor.coef. Fo:Fc: 0.972 / Cor.coef. Fo:Fc free: 0.966 / SU B: 1.627 / SU ML: 0.057 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.075 / ESU R Free: 0.077 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : REFINED INDIVIDUALLY
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 166.6 Å2 / Biso mean: 30.029 Å2 / Biso min: 12.72 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.54→31.47 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.54→1.58 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj










